上海藥物所開發抗新冠肺炎藥物靶標預測及虛擬篩選網絡應用平台
None
據中國科學院上海藥物研究所網站消息,近日,《藥學學報》英文刊(Acta Pharmaceutica Sinica B)發表了中科院上海藥物研究所開發的基於互聯網的抗新冠肺炎(COVID-19)藥物靶標預測及虛擬篩選平台(D3Targets-2019-nCoV)的介紹文章。該平台既可以為活性化合物預測潛在的作用靶標,也可開展基於多靶標多位點的藥物虛擬篩選,有望推動抗COVID-19藥物的快速研發。

由於新冠病毒(SARS-CoV-2)的感染和複製過程不僅依賴於病毒自身蛋白質,也涉及一系列宿主蛋白質,因此這些蛋白質都有可能成為開發抗新冠肺炎的藥物靶標。為此,科研人員一方面根據已公佈的SARS-CoV-2全基因組序列,利用生物信息學、同源建模、PDB庫下載等方法,獲得了20種新冠病毒自身編碼的蛋白質的三維結構;另一方面通過文獻調研收集了22種與病毒侵入、複製和釋放相關的人源蛋白,並通過PDB庫下載、同源建模、從頭建模等方法獲得了三維結構。
在獲得的42個蛋白質的三維結構基礎上,研究人員進一步運用數據庫搜尋、分子動力學模擬、潛在藥物結合位點預測等方法獲得了這42個蛋白質的69個不同的構象和 557個可能的藥物結合口袋,並由此開發出了D3Targets-2019-nCoV網絡應用平台(圖1)。該平台主要有兩個功能:一是針對在體內外實驗研究中發現的活性化合物,預測其可能的靶標蛋白,幫助理解藥物的作用機制,以促進結構優化和新藥研發;二是可以選擇與病毒侵入、複製和釋放相關的病毒蛋白和人源蛋白靶標,開展多靶標、多構象、多結合位點的藥物虛擬篩選,預測潛在的抗COVID-19活性化合物,以獲得作用機制更加豐富的先導化合物。
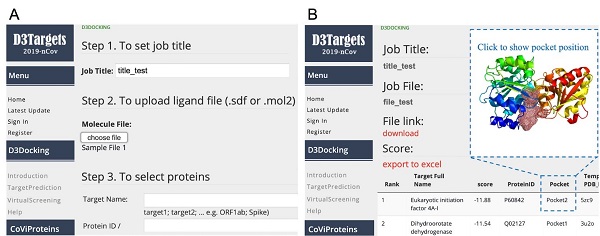
圖1. D3Targets-2019-nCoV的輸入界面(A)和輸出界面(B)
該平台具有多構象、多位點分子對接的獨特功能,這得益於研究團隊長期從事的基礎研究積累。多構象的產生使用了他們開發的高效分子動力學模擬新方法NUMD(J. Phys. Chem. B, 2013)及vsREMD(Biophys. J., 2020),這種方法能在蛋白質構象空間高效採集可用於藥物研發的可藥性構象(圖2)。蛋白質上潛在藥物結合位點的探索則運用了團隊發展的成藥性結合位點預測新方法D3Pockets(J. Chem. Inf. Model.,2019),可對蛋白質在構象轉變過程中的口袋變化進行分析,以探尋具有高穩定性、強相關性的正構或別構口袋(圖3)。這些新方法的應用成功地實現了多靶標、多構象、多位點的對接策略,預期可降低對接結果的假陰性率,提高藥物發現的成功率。
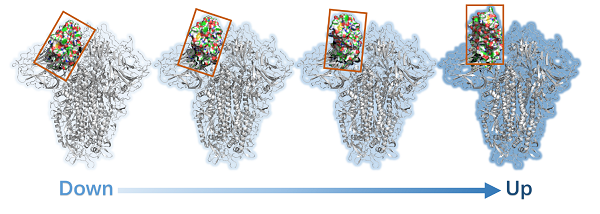
圖2. NUMD方法預測的S蛋白的多構象結果
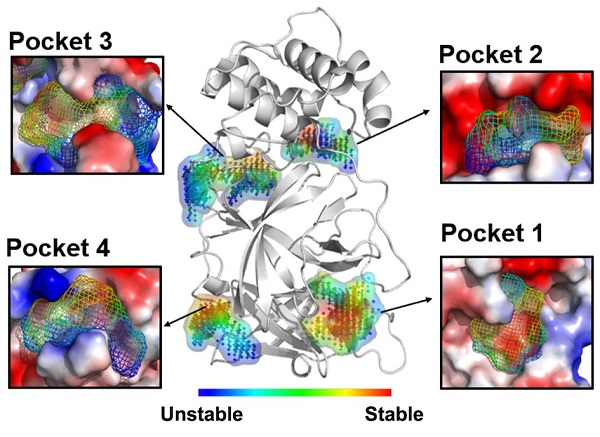
圖3. SARS-CoV-2 主蛋白酶上口袋的穩定性
該平台具有多方面的功能應用場景,如:(1)多靶標藥物的靶標預測;(2)老藥/活性化合物的靶標預測;(3)靶向特定蛋白質的多構象虛擬篩選;(4)化合物與指定靶標的結合模式預測;(5)用户可以從網站上免費下載蛋白模型,用於進一步的分子動力學模擬、藥物設計、構效關係研究等。同時,研究團隊認為,只根據最高對接打分來判斷靶標並不總是可靠的,需要綜合考慮對接打分、對接結合模式和蛋白功能等多方面因素,從而選出最可能的結果。該團隊也開展了應用研究,預測奈非那韋可能具有抗新冠病毒的活性(https://www.biorxiv.org/content/10.1101/2020.01.27.921627v1)。
D3Targets-2019-nCoV網站會在未來定期更新其靶標蛋白、蛋白質構象、可藥性結合位點等數據,用户可免費訪問D3Targets-2019-nCoV網站,註冊並提交任務。截止2020年5月3日,該網站已有超過9000的瀏覽量,共計1400多個訪客,分佈於全球50多個國家和地區。
該項工作最早在今年2月11日發佈於預印本網ChemRxiv(https://doi.org/10.26434/chemrxiv.11831163.v1),並在近期正式發表於Acta Pharmaceutica Sinica B(DOI: 10.1016/j.apsb.2020.04.006),題為D3Targets-2019-nCoV: a webserver for predicting drug targets and for multi-target and multi-site based virtual screening against COVID-19。論文第一作者為中科院上海藥物研究所研究生石禹龍,共同第一作者為張鑫賁、穆凱潔和彭誠,通訊作者為徐志建副研究員和朱維良研究員。
D3Targets-2019-nCoV網站地址:https://www.d3pharma.com/D3Targets-2019-nCoV/index.php
原文鏈接:https://doi.org/10.1016/j.apsb.2020.04.006