SARS-CoV-2校對酶識別錯配的結構基礎_風聞
观察员2号-2021-07-28 13:44
抽象的冠狀病毒 3’-5’ 外切核糖核酸酶 (ExoN) 位於非結構蛋白 (nsp) 10-nsp14 複合物中,通過校對 RNA 合成來提高複製保真度,並且對病毒生命週期至關重要。ExoN 還識別並切除整合到新生 RNA 中的核苷酸類似物抑制劑,從而削弱基於核苷酸類似物的抗病毒藥物的有效性。在這裏,我們展示了野生型和突變型 SARS-CoV-2 nsp10-nsp14 與帶有 3’ 端錯配的 RNA 底物複合物的冷凍電子顯微鏡結構,分辨率範圍為 2.5 Å 至 3.9 Å。這些結構揭示了 ExoN 底物特異性的分子決定因素,並深入瞭解了冠狀病毒 RNA 合成過程中錯配校正的分子機制。我們的研究結果為合理設計改進的抗冠狀病毒療法提供了指導。
SARS-CoV-2 是 COVID-19 大流行的病原體,已感染超過 1.6 億人,並導致全球超過 300 萬人死亡 ( https://covid19.who.int )。雖然現在有幾種 SARS-CoV-2 疫苗可用 ( 1 ),但沒有高效的抗病毒藥物來治療這種疾病。SARS-CoV-2 最重要的藥物靶點之一是其複製/轉錄複合體 (RTC),這是一種多亞基機器,可進行病毒基因組複製和轉錄,並在病毒生命週期中發揮重要作用 ( 2 , 3)。冠狀病毒 RTC 的核心是核心 RNA 依賴性 RNA 聚合酶 (RdRp)、nsp12 ( 4 ) 和兩個相關的輔助蛋白 nsp7 和 nsp8 ( 5)。SARS-CoV-2 RdRp 是核苷酸類似物抗病毒藥物(例如瑞德西韋)的一個有前景的靶標 ( 6 , 7 )。但是,核苷酸類似物抑制劑對冠狀病毒的RdRp的效力是由病毒nsp14的存在損害核糖核酸外切酶(外顯子)(8,9),一個RNA校對特有冠狀和型Nidovirales順序和關鍵的一些其他密切相關的病毒家族保持其異常大的 RNA 基因組的完整性 ( 9 – 11)。另外,從冠狀病毒等RNA病毒外顯子通過降解,否則將被宿主病原體識別受體(識別病毒雙鏈RNA(dsRNA的)中間發揮的宿主免疫應答逃避了重要作用12 - 15)。
Nsp14是一個雙功能酶,其既港口的3’-5’外顯子和mRNA帽鳥嘌呤-N7甲基(N7-轉移酶)活性(16,17)(圖1A)。nsp14 的 N 端外顯子結構域通過從新生 RNA 中去除錯誤摻入的核苷酸或核苷酸類似物來提高 RNA 合成的保真度,而 C 端 N7-MTase 結構域參與病毒基因組和亞基因組信使的 5’ 加帽過程RNA ( 16 – 18 )。nsp14 的 ExoN 活性受到 nsp10 的刺激,nsp10 與 ExoN 結構域結合併有助於穩定 ExoN 活性位點的結構。 18)。先前對 SARS-CoV nsp10-nsp14 複合物的研究將 nsp14 ExoN 域定義為 DED/EDh 型外切核酸酶,並通過結構比較和誘變分析確定了五個活性位點殘基 ( 19 )。然而,冠狀病毒 nsp10-nsp14 ExoN 與底物結合的分子細節仍不清楚。此外,病毒 ExoN 如何識別和切除新合成 RNA 3’ 端錯誤摻入的核苷酸或核苷酸類似物抑制劑,我們知之甚少。
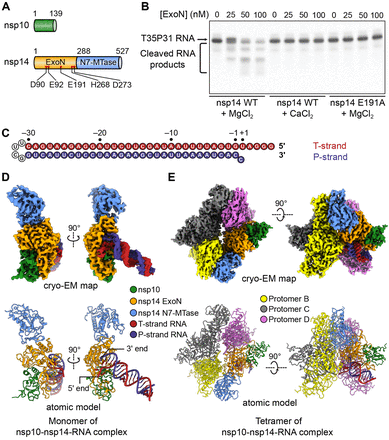 下載高分辨率圖像 在新標籤頁中打開 下載幻燈片圖 1 SARS-CoV-2 nsp10-nsp14-RNA 複合物的結構和功能概述。
下載高分辨率圖像 在新標籤頁中打開 下載幻燈片圖 1 SARS-CoV-2 nsp10-nsp14-RNA 複合物的結構和功能概述。
( A ) SARS-CoV-2 nsp10 和 nsp14 的域組織。域邊界殘基被編號。nsp14 ExoN 域中的五個催化殘基用紅點表示並標記。( B ) SARS-CoV-2 nsp10-nsp14 ExoN 複合物對 T35P31 RNA 底物的切割。標明瞭野生型 (WT) 或 E191A 突變型 ExoN 的濃度(以 nM 為單位)。RNA 通過變性聚丙烯酰胺凝膠電泳 (PAGE) 解析並通過 SYBR Gold 染色。顯示了三個生物學重複的代表性結果。(C ) 用於生化表徵和結構測定的 T35P31 RNA 底物的序列和編號。T-鏈,模板鏈;P鏈,產品鏈。T 鏈和 P 鏈 RNA 由 UUCG 四環連接。( D) SARS-CoV-2 nsp10-nsp14-RNA複合物單體形式的冷凍電鏡圖和原子模型。( E ) SARS-CoV-2 nsp10-nsp14-RNA複合物四聚體形式的冷凍電鏡圖和原子模型。
為了理解 SARS-CoV-2 ExoN 的底物識別和催化機制,我們構建了一個髮夾 RNA 底物(以下簡稱 T35P31),其中包含一條模板鏈(T 鏈,也是 ExoN 的非易剪鏈)三個起始鳥苷,然後是 SARS-CoV-2 基因組的 3’-末端 32 個核苷酸 (nt)(不包括 poly(A) 尾)和一個 31-nt 產物鏈(P-鏈,也是易裂解的ExoN 鏈)以胞苷-5’-單磷酸(CMP)結尾,導致 3’ 末端的 CU 錯配(圖 1C)。預先形成的 SARS-CoV-2 nsp10-nsp14 複合物在 MgCl 2存在下消化 T35P31 RNA 底物(圖 1B)。為了獲得穩定的 nsp10-nsp14-RNA 複合物,我們用 MgCl 2使用重構緩衝液中的CaCl 2,或將 ExoN 活性位點突變 E191A 引入到 nsp14。兩種措施都保留了 RNA 結合能力,但取消了 nsp10-nsp14 複合物的 RNA 切割活性(圖 1B和圖S1 和 S2)。
重組的 nsp10-nsp14-RNA 複合物通過尺寸排阻色譜 (SEC) 進行純化,並通過單顆粒冷凍電鏡進行分析。WT 和突變體 nsp10-nsp14-RNA 複合物的最終冷凍電鏡圖分別精製為 3.9 Å(圖 S1 和 S3)和 3.4 Å(圖 S2 和 S3)。除了 RNA 底物和蛋白質殘基側鏈構象的微小差異外,這兩種結構幾乎相同,所有蛋白質 Cα 原子的 RMSD 為 0.39 Å(圖 S4)。ExoN 活性位點位於 nsp14 ExoN 結構域並由 nsp10 的 N 末端支持,結合 RNA 的 3’ 端,將其與 5’ 突出端分開(圖 1D)。大多數 RNA 螺旋在溶劑暴露空間中仍可自由進入(圖 1D)。
為了探索 SARS-CoV-2 RdRp 和 ExoN 之間可能的聯繫,SARS-CoV-2 nsp8 被包含在複合物的重構中,並被發現與 SEC 柱上的 nsp10-nsp14-RNA 複合物共洗脱(圖 S1A 和 S2A)。然而,它沒有與冷凍電鏡樣品中的 nsp10-nsp14-RNA 形成穩定的複合物,並且僅在一小部分顆粒中觀察到(圖 S2C),表明 nsp8 與 nsp10-nsp14-RNA 的關聯複雜是弱的和動態的。儘管對 nsp8 結合類的進一步計算機分類沒有產生具有 nsp8 高分辨率特徵的圖,但 6 Å 低通過濾圖顯示出沿着 RNA 雙鏈體溶劑暴露區域的強大額外密度(圖 S2C) . 當從 SARS-CoV-2 RdRp 複合結構中對接 nsp8 時 ( 20) 作為剛體進入密度,其 N 端延伸的螺旋通常很好地擬合,並且其相對於 RNA 骨架的方向與 SARS-CoV-2 RdRp 複合物 ( 20 , 21 ) 中的方向相匹配(圖 S5A)。對接將 nsp8 的 C 端結構域置於冷凍電鏡密度之外,但與 N 末端螺旋相鄰的冷凍電鏡密度未被佔用(圖 S5A),表明 nsp8 可能採用與處於RdRp 複合體。這與之前的結構研究一致,證明了 nsp8 ( 21 – 23)。nsp8 與 nsp10-nsp14-RNA 複合物的結合模式表明 nsp8 可能有助於穩定底物結合以進行外顯子介導的 RNA 切割。事實上,外切核糖核酸酶活性測定顯示 nsp8 增強了 nsp10-nsp14 複合物的 RNA 消化(圖 S5B)。作為 ExoN 和 RdRp 複合物中的常見成分,nsp8 可能在兩種酶之間的 RNA 底物轉移中發揮作用。然而,nsp8在體內錯配校正中的詳細功能需要進一步研究。
使用突變 ExoN 重組的冷凍電鏡樣本包含一個代表 nsp10-nsp14-RNA 複合物四聚體形式的類(圖 1E和圖。S2C)。四聚將 3D 重建的分辨率提高到 2.5 Å,而不會影響複合物的結構(圖 S2C 和 S6A)。然而,nsp10-nsp14-RNA 複合物的四聚化可能會阻止 nsp8 結合(圖 S6B)。因此,在四聚體圖中未觀察到沿 RNA 雙鏈體的 nsp8 樣密度。儘管來自 WT nsp10-nsp14-RNA 複合物數據集的 2D 類平均值也揭示了可能代表複合物四聚體形式的粒子(圖 S1B),但此類粒子的數量有限,無法進行有意義的 3D 重建。除非另有説明,我們將使用 nsp10-nsp14-RNA 複合物的四聚體形式進行 ExoN 活性位點的後續結構分析及其與 RNA 底物的相互作用,因為其分辨率更高。
與 SARS-CoV nsp10-nsp14 複合物的 apo 形式 ( 19 ) 相比,SARS-CoV-2 WT nsp10-nsp14-RNA 複合物的結構顯示出 α4-α5 和 α2-α3 環的局部構象變化,導致稍微變窄的 RNA 結合袋(圖 2A)。底物結合也會導致 ExoN 活性位點的完全組裝。雖然 apo ExoN 僅捕獲一種二價金屬離子 ( 19 ),但與 RNA 結合的 ExoN 在其催化中心包含兩個金屬離子結合位點(圖 2B和圖 S7A)。金屬離子 A 由 D90、E92 和 D273 的羧酸氧配位,激活水分子進行親核攻擊。金屬離子 B 由 D90 和 E191 配位並穩定 –1C P的 O3’ 離去基團(核苷酸編號示於圖1C )(圖2B和圖S7A)。在 E191A 突變體 nsp10-nsp14-RNA 複合物中,由於缺少 E191 側鏈羧酸鹽,金屬離子 B 的配位很差,並且與 –1C P的 O3’ 離去基團的配位距離之外(圖 2C和圖2C)。 S7B)。第五催化渣油H268,其功能是作為一般鹼和磷酰基轉移反應過程中去質子化的催化水(24,25)(圖2B和圖S7A),位於nsp14α4-α5環和移位2.6埃朝向易裂解的磷酸鹽,在存在 RNA 底物的情況下完成活性位點(圖 2A)。
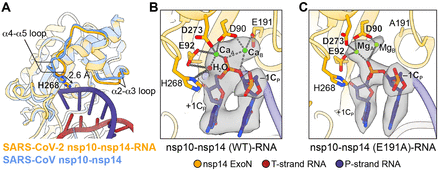 下載高分辨率圖像 在新標籤頁中打開 下載幻燈片圖2 SARS-CoV-2 ExoN的活性位點構象和催化機制。
下載高分辨率圖像 在新標籤頁中打開 下載幻燈片圖2 SARS-CoV-2 ExoN的活性位點構象和催化機制。
( A ) SARS-CoV nsp10-nsp14 複合物(矢車菊藍,PDB 5C8U)和 SARS-CoV-2 nsp10-nsp14-RNA 複合物(橙色)的疊加説明了 α2-α3 和 α4-α5 環的構象變化和 2.6 Å 底物結合後 H268 向 RNA 移動。( B ) SARS-CoV-2 nsp10-nsp14 (WT)-RNA複合物的活性位點結構。Ca 2+離子,綠色球體;催化水,紅色球體。P 鏈 RNA 中的核苷酸殘基用下標“P”表示。+1C P、–1C P、催化水和兩個活性位金屬離子疊加在一起,它們的冷凍電鏡密度輪廓為 10σ。( C ) SARS-CoV-2 nsp10-nsp14 (E191A)-RNA複合物的活性位點結構。鎂2+離子,綠色球體。+1C P、–1C P和兩個活性位金屬離子疊加在一起,它們的冷凍電鏡密度輪廓為 7σ。
沙粒病毒科拉沙病毒 (LASV) 的核蛋白 (NP) 代表了在 RNA 病毒中發現的唯一其他外顯子組 ( 14 , 15 , 26 )。儘管冠狀病毒 nsp14 和沙粒病毒 NP 已經進化出不同的附加域來解決不同的功能 ( 15 ),但它們的外顯子域的整體摺疊和活性位點構象是相似的(圖 S7、C 和 D)。主要區別在於 LASV NP 的 D466 承擔了 nsp14 中 E191 的作用,以配位金屬離子 B,可能是由於其較短的側鏈而通過中間水分子(圖 S7D)。
淺層 SARS-CoV-2 ExoN 底物結合袋僅包含 dsRNA 的鹼基對 (bp)–1 和–2,通過 nsp14 中的 nsp10 和 K9、W186 和 Q245 的 A1 與 RNA 骨架相互作用(圖 3A)。在dsRNA基板的3’末端,nsp14分離不匹配的一對CU和翻轉+ 1U Ť出RNA雙螺旋(圖3,A和B)。其結果是,在SARS-CoV的-2外顯子的活性位點結合是被1-nt的3’突出端,其包括+ 1C的dsRNA P(圖3,A和B)中,從在其他RNA病毒中觀察到的襯底結構不同和校對 DED/EDh 核酸外切酶 ( 26 – 28 ) 和先前預測的 SARS-CoV ExoN ( 8, 18 )。SARS-CoV-2 ExoN 的底物特異性是由 nsp14 和 RNA 底物之間的許多相互作用促成的(圖 3,A 和 B)。SARS-CoV-2 ExoN 底物結合口袋底部的 F146 與 3’ 端未配對的 +1C P堆疊在一起。N104插入到dsRNA的小溝,並建立與-1G的核苷鹼基和2’-OH基團兩個氫鍵Ť分別。H95 與未配對的 +1C P大致共面,與胞苷鹼基氫鍵合併與 –1G T堆疊(圖 3B)。H95 作為氫鍵供體和受體的能力可能允許它容納所有四種類型的核苷酸,解釋了 nsp14 對底物序列的相對不敏感性 ( 18 )。由於打破該鹼基對所需的能量更高,SARS-CoV-2 ExoN 對 dsRNA 底物的消化可能會在 CG 鹼基對處變慢。此外,位於 ExoN RNA 結合口袋邊緣的 P142 與 H95 一起工作以限制 T 鏈側底物結合口袋的深度,並可能迫使 RNA 底物 3’-末端的鏈分離CU 不匹配對(圖 3,B 和 C)。用於分離錯配鹼基對的較低能量可以解釋冠狀病毒 ExoN 對 3’ 端錯配的 dsRNA 底物的偏好,而不是完美匹配的底物 ( 18 )。相比之下,LASV ExoN RNA 結合口袋在非易裂鏈側有一個稍深的開口,因此能夠容納完全鹼基配對的 dsRNA 底物(26)(圖 3D)。這與其作為 dsRNA 降解免疫抑制劑的作用一致,而不是 RNA 合成校對者 ( 14 , 15 )。在光譜的另一端,是與 DNA 聚合酶相關的校對外顯子,例如大腸桿菌DNA 聚合酶 III (Pol III) ε 亞基。它有一個更窄的 DNA 結合口袋,部分原因是它與 Pol III α 亞基緊密結合,並且只能適合單鏈 DNA 底物(27)(圖 3E)。nsp14 中所有與 RNA 接觸的殘基在不同冠狀病毒屬之間都高度保守(圖 S8),表明冠狀病毒 ExoN 具有共享的 RNA 底物識別機制。
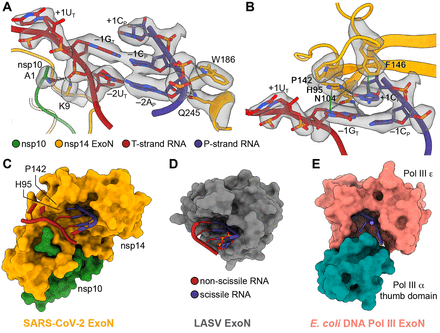 下載高分辨率圖像 在新標籤頁中打開 下載幻燈片圖3 SARS-CoV-2 ExoN識別底物的機制。
下載高分辨率圖像 在新標籤頁中打開 下載幻燈片圖3 SARS-CoV-2 ExoN識別底物的機制。
( A ) SARS-CoV-2 nsp10-nsp14 ExoN 和 T35P31 RNA 骨架之間的相互作用。T鏈RNA中的核苷酸殘基用下標“T”表示,P鏈RNA中的核苷酸殘基用下標“P”表示。氫鍵和鹽橋顯示為灰色虛線。相互作用的核苷酸和蛋白質殘基與其輪廓為 7σ 的冷凍電鏡密度疊加。( B ) SARS-CoV-2 nsp10-nsp14 ExoN 和 T35P31 RNA 在 +1 和 –1 核鹼基位置之間的相互作用。氫鍵和鹽橋顯示為灰色虛線。π-π 堆積相互作用用綠色虛線表示。相互作用的核苷酸和蛋白質殘基與其輪廓為 7σ 的冷凍電鏡密度疊加。( C) SARS-CoV-2 nsp10-nsp14 ExoN 底物結合口袋的表面表示顯示了 T 鏈側的受限開口,阻止了底物 RNA +1 位置的鹼基配對。為清楚起見,未顯示 nsp14 的 N7-MTase 域。( D ) LASV NP ExoN 域的表面表示。完全鹼基配對的 dsRNA 底物(如圖所示)結合在 LASV ExoN 的底物結合口袋中。( E )大腸桿菌DNA Pol III ExoN 複合物的表面表示。狹窄的底物結合口袋只允許 ssDNA 進入。
作為 3’-5’ 外切核糖核酸酶,SARS-CoV-2 nsp10-nsp14 特異性識別 3’-末端核苷酸的 2’-和 3’-OH 基團。+1C P的 2’-OH分別與 H95 和 G93 的羰基氧形成兩個氫鍵,而核苷酸的 3’-OH 與 G93 主鏈氮和催化殘基 E92 形成氫鍵(圖 4A))。為了檢查 3’-末端核苷酸的 2’-和 3’-OH 基團對 SARS-CoV-2 ExoN 的 RNA 切割效率的影響,我們使用 32-nt 單鏈 RNA (ssRNA) 進行了核酸外切酶測定底物(稱為 P32 RNA)以標準核糖核苷酸或在 2’ 或 3’ 位置修飾的核苷酸結尾(圖 4B))。SARS-CoV-2 nsp10-nsp14 有效地切割未修飾的 ssRNA,儘管需要顯着更高的酶濃度才能獲得與在具有相同 P 鏈序列的 dsRNA 底物上實現的切割相當的切割(圖 S9)。這可能是由於 ssRNA 與 SARS-CoV-2 ExoN 的結合較弱,這是由於 T 鏈側蛋白質-RNA 相互作用的喪失導致的(圖 3,A 和 B)。ExoN 接受 ssRNA 和 dsRNA 底物的能力表明體內錯配校正的兩種可能模式。ExoN 可能結合並切割由 RdRp 回溯導致的 P 鏈 RNA 的 3’-末端單鏈區域,如先前研究所提出的 ( 21 , 29)。或者,包含 3’ 端錯配的 dsRNA 底物可能會從 RdRp 上分離,隨後被 ExoN 識別以進行錯配切除。
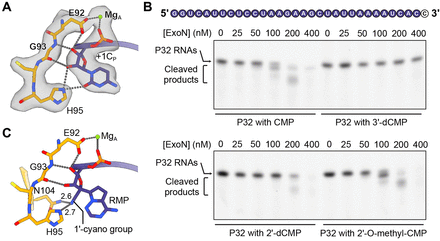 下載高分辨率圖像 在新標籤頁中打開 下載幻燈片圖 4 抗病毒設計的結構洞察。
下載高分辨率圖像 在新標籤頁中打開 下載幻燈片圖 4 抗病毒設計的結構洞察。
(A)SARS-CoV-2 nsp10-nsp14 ExoN與其2’-和3’-OH基團介導的3’-末端核苷酸之間的相互作用。Mg 2+離子,綠色球體。氫鍵顯示為灰色虛線。+1C P及其相互作用的蛋白質殘基與輪廓為 7σ 的冷凍電鏡密度疊加。( B ) SARS-CoV-2 nsp10-nsp14 複合物對各種 P32 ssRNA 底物的裂解。在其 2’- 或 3’-OH 基團上帶有不同修飾的 3’-末端 CMP 顯示為白色圓圈中的黑色字母。顯示了 ExoN 的濃度(以 nM 為單位)。RNA 通過變性聚丙烯酰胺凝膠電泳 (PAGE) 解析並通過 SYBR Gold 染色。顯示了三個生物學重複的代表性結果。( C) 預測了 SARS-CoV-2 nsp10-nsp14 ExoN 與單磷酸瑞德西韋 (RMP) 之間的相互作用,該相互作用以 P 鏈 RNA 的 3’-末端 +1 位置為模型。
在測試的酶濃度範圍內,去除 3’-末端核苷酸的 2’-或 3’-OH 基團可以減少或幾乎消除 SARS-CoV-2 ExoN 的核酸水解降解(圖 4B),與之前的發現一致SARS-CoV ExoN ( 18 ) 並反映了 2’- 和 3’-氧在冠狀病毒 ExoN 催化中的重要作用。另一方面,3’-末端胞苷的 2’-O-甲基化不會顯着影響 SARS-CoV-2 nsp10-nsp14 對底物的切割(圖 4B),可能是因為 2’-氧之間的一些相互作用和 nsp14 被保留。
Remdesivir 是唯一獲得 FDA 批准的用於治療 COVID-19 的核苷酸類似物抗病毒藥物。為了評估瑞德西韋是否可以被 SARS-CoV-2 ExoN 有效切除,我們在 P 鏈的 +1 位置模擬了抑制劑的摻入形式,即單磷酸瑞德西韋(RMP)(圖 4C)。建模的 RMP 保留了 nsp14 和 3’-末端 CMP 之間形成的大部分有利相互作用。此外,RMP 的 1’-氰基,其延遲 RdRp 停滯活性的決定因素 ( 6 , 7 ),緊貼在 H95 和 N104 之間的空間中,並與來自兩個殘基的側鏈氮原子形成氫鍵(圖 4C )。這些觀察結果表明,含有 RMP 的產物 RNA 可能是冠狀病毒 ExoN 的底物,這與以 RMP 終止的 RNA 對 ExoN 切除沒有顯着抗性的發現一致(30)並且缺乏 ExoN 校對活動的冠狀病毒對瑞德西韋明顯更敏感( 31 )。
我們的研究深入瞭解了 SARS-CoV-2 RNA 合成過程中錯配校正的機制,並揭示了對 ExoN 識別和催化至關重要的底物結構特徵,為特定且有效的 ExoN 抑制劑的結構引導設計提供了基礎。將此類 ExoN 抑制劑與基於核苷酸類似物的病毒 RdRp 抗病毒藥物共同給藥可能構成對 COVID-19 的更有效治療。此外,我們的研究揭示了 ExoN 抗性核苷酸類似物抑制劑的開發。特別是,我們表明 RNA 底物的遊離 3’-OH 對於 ExoN 的核酸外切降解至關重要。已經表明,3’-脱氧核糖核苷酸可以通過來自其他正鏈 RNA 病毒(如 HCV 和脊髓灰質炎病毒)的 RdRp 有效地整合到新生的 RNA 中,32 , 33)。因此,3’-脱氧核苷酸類似物有可能作為有效的冠狀病毒 RdRp 鏈終止子,也能抵抗 ExoN 切除。儘管如此,核糖環上其他位置的修飾也值得進一步探索。