AI設計蛋白質新方法登Science!利用強化學習,直接根據預想優化結構_風聞
量子位-量子位官方账号-04-21 21:46
明敏 發自 凹非寺
量子位 | 公眾號 QbitAI
現在,強化學習不僅能下圍棋,還能設計蛋白質了!
RoseTTAFold團隊獨創性方法,利用強化學習、自上而下設計蛋白質複合物結構,登上最新一期Science。

利用該方法,團隊設計出了上百個蛋白質結構。
經電子顯微鏡等觀測證實,這些結構大部分都能在實驗室中合成。
而且預期納米結構和實際納米結構之間的平均偏差小於單個原子直徑,即實現了精確原子級設計。
在實際應用上,由該方法設計出的蛋白質能有效在小鼠體內產生有用抗體,併為未來開發出更有效的疫苗和藥物創造可能。
因此團隊表示,這是AI設計蛋白質領域的一個里程碑式研究,或將為蛋白質設計領域開創新時代。
作為AI設計蛋白質領域的明星團隊,華盛頓大學David Baker教授曾率隊開發出精度上可媲美AlphaFold2的蛋白質預測設計方法RoseTTAFold。
並在實際計算中達到了更快更輕便的效果,只需要一個英偉達RTX2080 GPU,10分鐘就能算出蛋白質結構。
先給預設再不斷優化
強化學習的原理,就是通過不斷地試錯學習,讓agent(智能體)通過與環境進行交互獲得獎賞指導行為,目標是獲得最大的獎賞。
將它應用到蛋白質設計方面,科學家們為程序提供了數百萬個簡單的起始分子,然後讓軟件進行一萬次嘗試,隨機地改善每一個預定目標。
程序將以特定方式拉長或彎曲蛋白質,直到學會如何將它們設計為所需的結構。
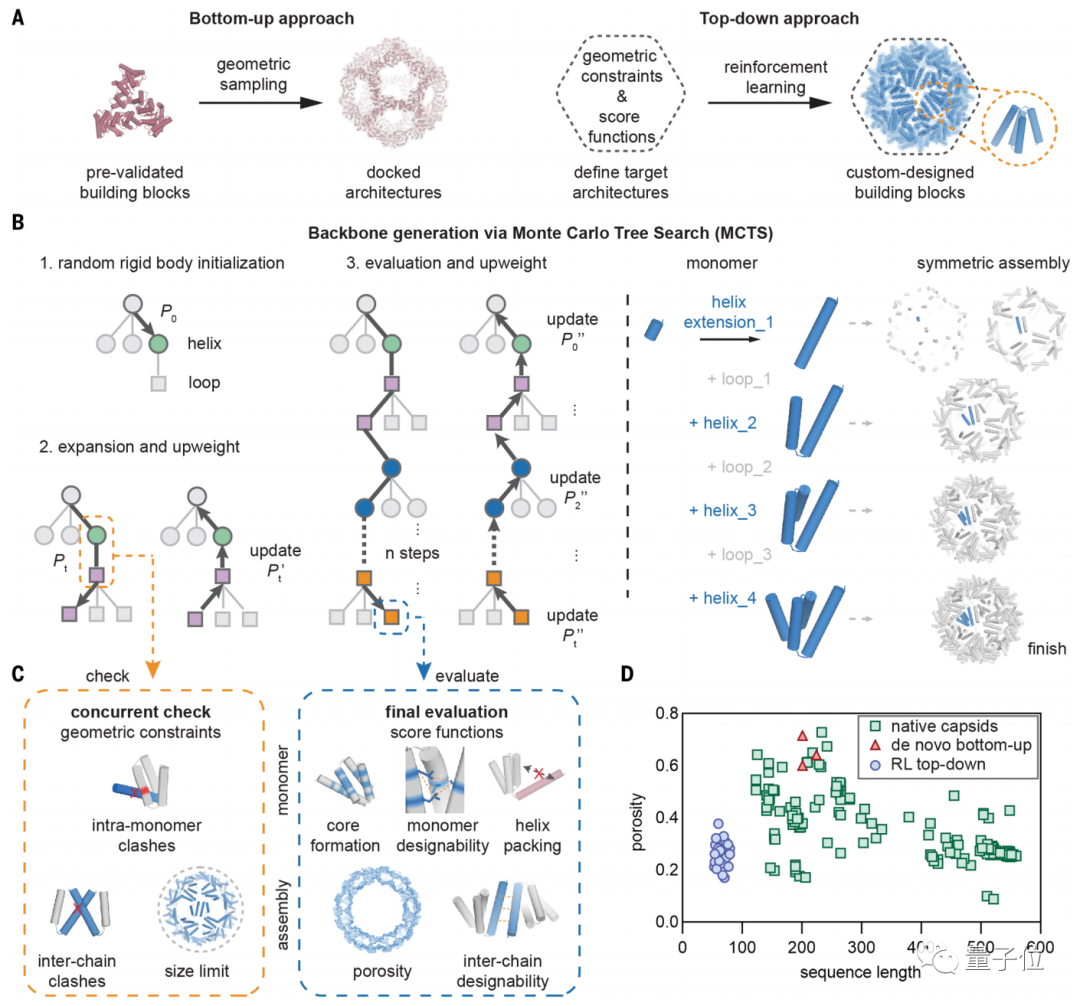
該方法先給出了先驗約束和得分函數定義目標結構,然後通過強化學習不斷探索,以得到定製化的構建模塊。
它有別於此前自下而上的設計方法。程序將不再侷限於依據已知的寡聚體原件來設計蛋白質,並能從全局角度來優化整個蛋白質結構。
然後再通過蒙特卡洛樹搜索算法,確定骨幹生成。
該方法能實現給出一個“遊戲狀態”並選擇勝率最高的下一步。AlphaGo中應用了這一原理。
在展開的每個階段,如果出現了違規情況就中止搜索樹;在模擬完成後,則通過一系列分數函數進行最終評估。
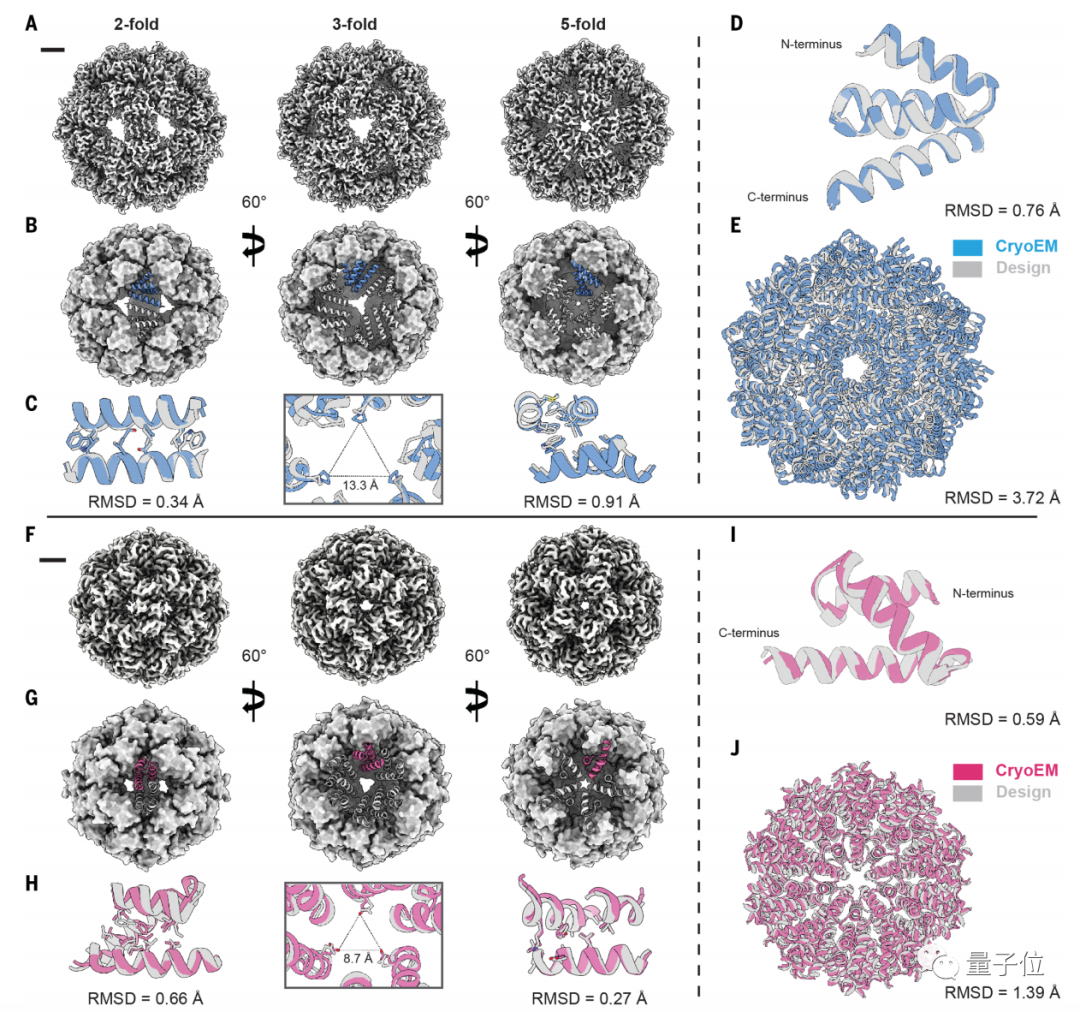
結果表明,面對傳統“自下而上”方法無法構建的蛋白結構,最新方法不僅能設計結構,而且結果和冷凍電鏡結構和計算機模型非常接近。
研究人員構建了數百種結構後表示,這些結構中的大多數都能在實驗室中構建。
而且團隊表示,這種方法不僅準確性高,還高度可定製化。
比如可以要求它設計出無孔隙、小孔或大孔不同要求的球狀結構,這種能力還有待進一步挖掘。
據瞭解,該團隊致力於設計由許多蛋白質分子組成的新型納米級結構。這需要設計蛋白質組件,並能夠讓納米結構自組裝。
在由AI設計的結構中,每個原子都出現了預定的位置上。即預期結構和可實現納米結構之間的平均偏差不到一個原子的直徑,這也可以被稱為精確原子設計。
由此團隊認為,這種方法能夠設計出藥物、疫苗等方面的蛋白。
華盛頓大學幹細胞和再生醫學研究所的研究人員使用血管細胞中的原代細胞(Primary cells)模型,證明了新方法設計的蛋白質結構優於此前方法。
比如在結構上構建出了更為聚集的信號接收受體,這提升了血管的穩定性。
團隊介紹
該研究來自華盛頓大學David Baker教授團隊。

David Baker現在是華盛頓大學蛋白質設計研究所所長。1984年在哈佛大學本科畢業,後赴加州大學伯克利分校攻讀博士學位。
他致力於研究蛋白質結構預測、設計以及蛋白質摺疊機制等領域。是利用AI探索蛋白質結構領域的大佬級人物。
最為人熟知的工作是RoseTTAFold。
這種蛋白質結構設計方法,不僅在精度上媲美AlphaFold2,並且在算法上更輕便。更有意思的是,它和AlphaFold2同天開源,一個登Sciecne,一個登上Nature。
去年,該團隊還提出了一種新的蛋白質設計策略,針對幾乎任何感興趣的靶點蛋白,RoseTTAFold都能設計出與之緊密結合的蛋白質分子作為候選藥物,應用可覆蓋癌症、新冠等諸多疾病。
而後該團隊發表在Science上的研究表明,機器學習可以比以前更快速和精確地構建蛋白質分子。
值得一提的是,在產業化方面David Baker以科學聯合創始人身份,加入了英國AI創新藥企CHARM Therapeutics,去年該公司獲得了5千萬美元融資。
論文地址:https://www.science.org/doi/10.1126/science.adf6591
參考鏈接:https://phys.org/news/2023-04-board-games-protein.html