母親的染色體端粒長度只有父親的37%?最新端粒測序技術問世,揭示父母本端粒差異_風聞
时光派-时光派官方账号-聚集全球前沿、全面、专业抗衰资讯1小时前

在過去很長一段時間裏,端粒都被視為生命的沙漏,或者一個抽象的尺子:我們每老一歲,它就會磨損一點。
而今年4月份發表在《Science》的重磅研究就發現,哪怕是同一個個體,不同染色體上的端粒長度可能也存在顯著差異。比如,染色體A的端粒可能會比染色體B的端粒長6000個bp(鹼基對,DNA的基本單位)[1],要知道,通常端粒總長最多也才不過10000bp!
精細化的測序讓人們知道,我們曾經對端粒長度的認知僅是冰山一角,那麼在染色體端粒長度差異的背後,又包含了什麼秘密呢?
最近,端粒研究領域的大神,美國沙克研究所的Jan Karlseder教授團隊百尺竿頭更進一步,利用他們最新開發的Telo-seq技術,深入研究發現,端粒長度背後竟還有這麼多奧秘[2]。


通常來説,端粒被我們視作細胞壽命的倒數計時器,每次細胞分裂時,端粒就會變短一點,當端粒變得過短時,細胞會停止分裂,進入衰老或凋亡階段。
儘管我們常説**“細胞衰老不等於個體衰老”,比如年輕個體體內同樣存在衰老細胞**,但他們的細胞衰老通常並非是由端粒縮短引起的,而主要是氧化應激[3]和紫外線照射[4]等其他因素導致的。因此,端粒的長度依舊是我們評估細胞乃至整個身體生理年齡的參考。
但是,準確測量端粒長度仍舊是一個不夠成熟的課題。傳統的短讀長測序通常只能測出50-300鹼基的序列,這對於一般的DNA序列來説還算夠用,但面對端粒這種包含大量重複序列的選手,就有些抓瞎了,我們用兩段文本來做比較,首先是一般DNA序列:

像這樣重複文字不多的序列,短讀長測序讀出的片段很容易通過上下文信息判斷出每一個片段的位置,也就比較容易準確地還原出原本的完整序列,而大量重複序列則是這樣的:
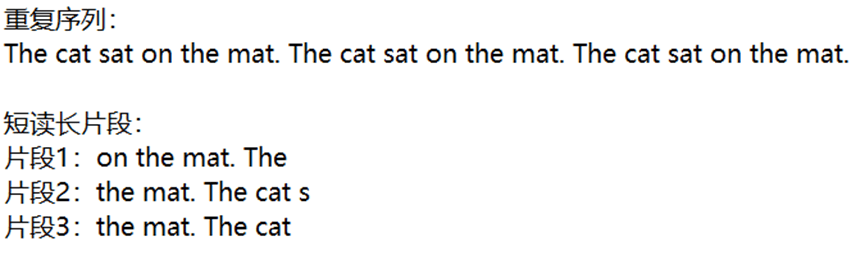
你會發現很難通過上下文確定三個片段之間的順序,因為這些短片段的內容在原文中出現了好幾次,所以面對大量重複序列,短讀長測序技術就很難拼湊出原本的完整序列,也就很難準確測量長度。
而為了解決這一難題,PacBio公司開發出了長讀長測序技術,可以一次性產生1k到2w個鹼基的讀長[5],有了更多的上下文信息,片段的拼接就容易多了,準確率也高了不少。
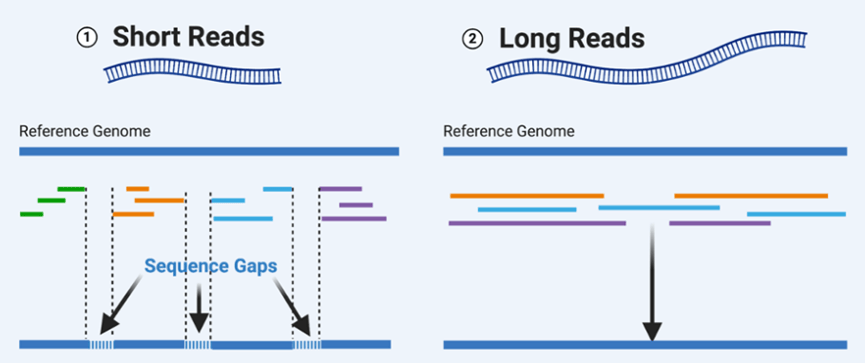
可是,長讀長測序固然強大,卻並不是專門為端粒設計的,它是用來測全基因組的,對於人類細胞來説,端粒僅佔全基因組的0.015%,直接用長讀長測序技術測端粒長度,無異於大炮打蚊子,不僅浪費還效率低下。因此,本文研究者們又開發了針對端粒的測序方式。

在Jan Karlseder教授團隊開發的新方法——Telo-seq中,DNA序列的端粒部分和非端粒部分可以被有效地分離,甚至能識別出每一段端粒來自哪條染色體。在此基礎上,再使用長讀長測序技術,就能精確地檢測出端粒序列了。
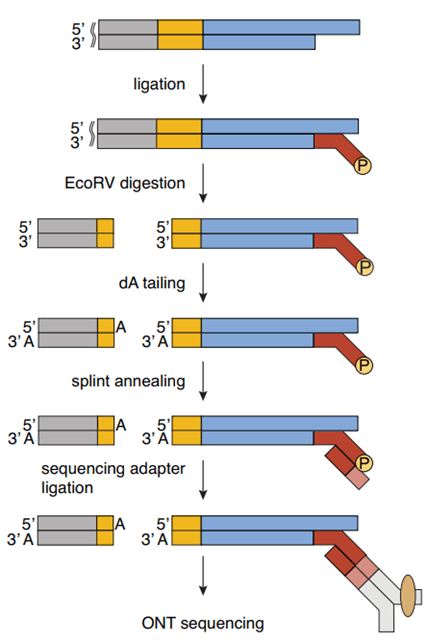
圖注:藍色代表端粒,灰色代表染色體,黃色代表端粒序列和一般染色體序列之間的過渡序列,而Telo-seq可以識別並切斷這種過渡序列
接下來,讓我們看看Telo-seq的效果如何。研究者測了B淋巴細胞系HG002的端粒長度,結果同樣發現了不同染色體臂之間的端粒長度差異,比如17號染色體的q臂端粒長度在3088-3139bp之間,21號染色體p臂端粒長度在9603-9835bp之間,相差高達6700bp!
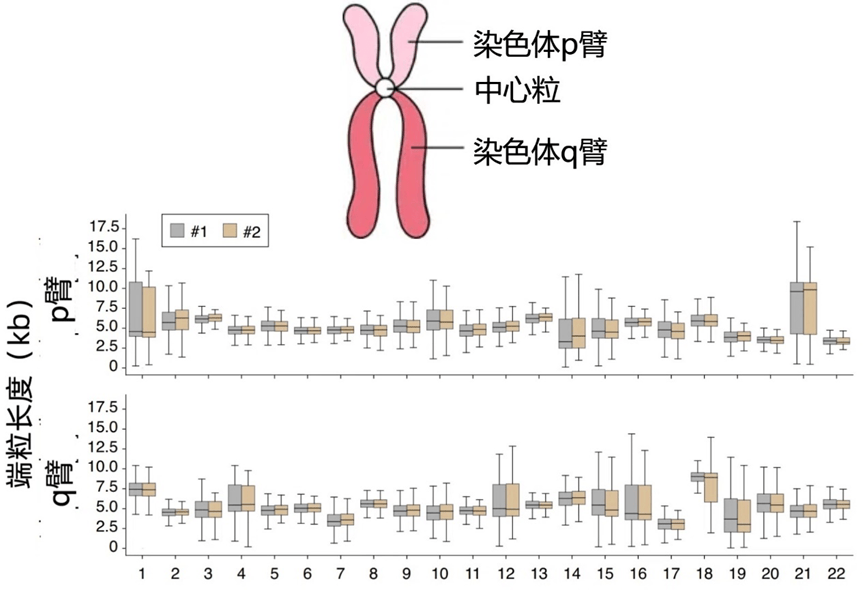
圖注:橫軸數字代表了人類體細胞的22條染色體,#1和#2代表了兩次重複實驗,而重複實驗的結果高度一致,意味着Telo-seq的測序結果穩定性較強
通常來説細胞每分裂一次,端粒就會丟失25-200bp[6],那麼接下來,我們來看看這種微小的變化是否也能被Telo-seq捕捉到。
研究者們在體外培養了表達人類乳頭瘤病毒E6和E7致癌基因(IMR90 E6E7,可以讓細胞突破海弗利克極限)的IMR90人肺成纖維細胞,並傳代多次,然後,他們用Telo-seq檢測了這些細胞在分裂66次、68次、71次、85次、96次和106次時的端粒長度。
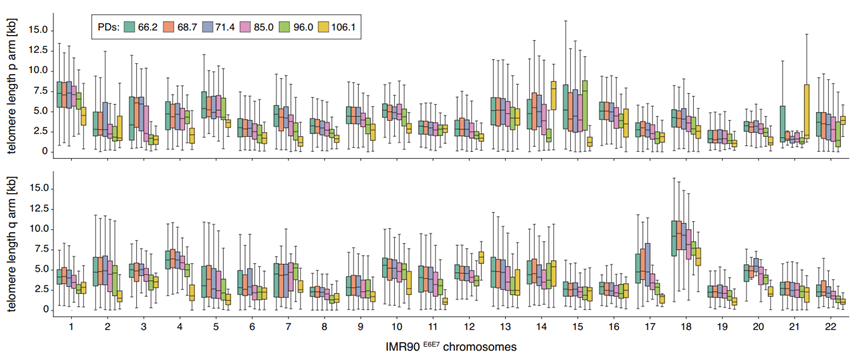
圖注:上半為染色體p臂,下半為染色體q臂,PDs為倍增次數,可以近似理解為細胞分裂次數
結果顯示,平均端粒長度從66次分裂時的4294bp,到106次分裂時的2746bp,端粒長度平均每分裂一次就縮短39bp。
如果説到此為止,Telo-seq也只是比肩“前人”的話,那麼接下來它的“絕活”就要登場了!
我們每個人的染色體有一半來自母親,一半來自父親,此前沒有人知道父本和母本染色體的端粒縮短速度是否一致,這次,Jan Karlseder教授給出了答案:
即使是同一條染色體,來自父親的端粒和來自母親的端粒也會在長度上大相徑庭,比如HG002細胞系中,1號染色體p臂的母本端粒長度為4165bp,而父本則長達11139bp。
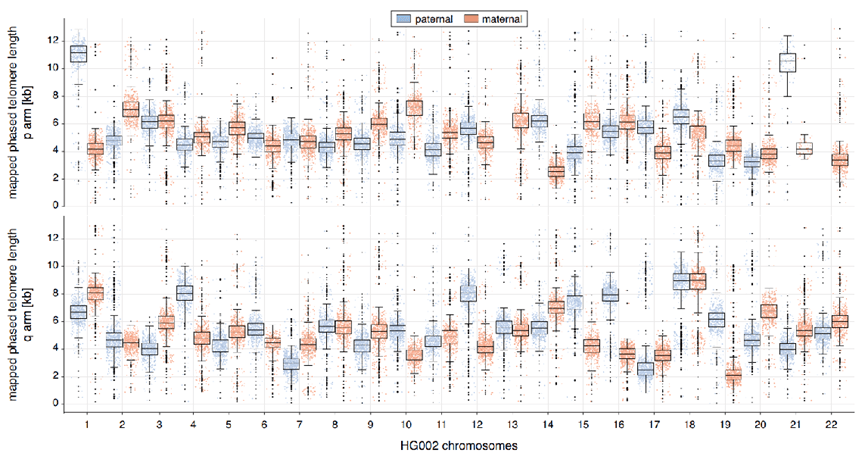
圖注:藍色為父本,紅色為母本
最後,研究者還對20到94歲捐贈者的成纖維細胞進行了Telo-seq分析,不出意外,總體趨勢顯示端粒長度與年齡成反比關係,然而,若仔細區分每條染色體,會發現相比於染色體因素,年齡因素帶來的端粒長度差異更小:
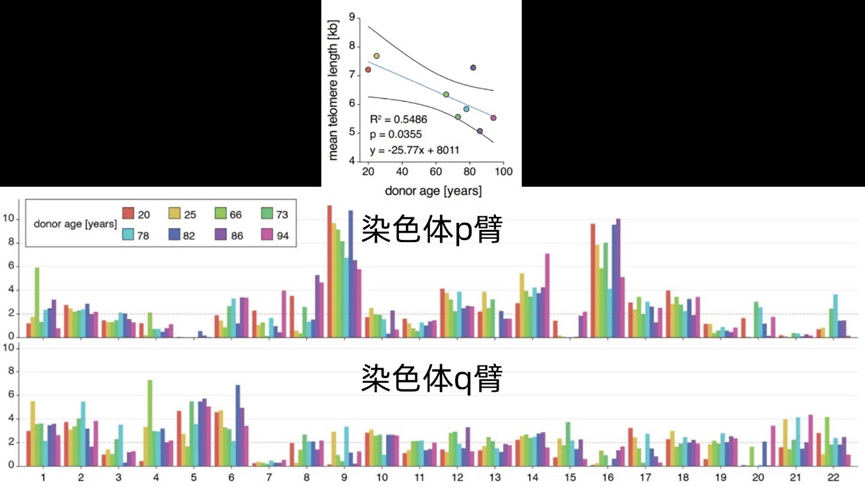
圖注:上圖為不區分染色體的情況下,不同捐贈者細胞的端粒平均長度隨年齡的變化趨勢;下圖不同顏色代表了不同的年齡,並展示了22條染色體的p臂和q臂的端粒長度
簡單總結一下,我們不難得出一些有意思的新發現:
1. 端粒長度受到染色體臂的影響比年齡更甚,就像組織器官的差異對基因表達的影響比年齡差異更大一樣,因此今後在對比端粒長度、端粒縮短速率時,或許該先控制好染色體臂這一變量,也許,同一號染色體臂之間的端粒長度對比,比整體端粒長度的對比更有意義。
2. 端粒長度在不同染色體臂之間的差異,不僅僅與染色體本身的特性有關,可能也受到同一條染色體中,父本與母本的染色體端粒長度差異的影響。

如果用傳統方法對端粒長度的測序是360p黑白照片的話,那麼Telo-seq下的端粒測序結果,就可以説是1080p高清全綵大圖了,不僅精度更高,而且還能區分染色體和父本母本,頓覺端粒長度背後的奧秘如此廣闊深邃。

既然科學家們在這項新技術加持下,能夠獲取大量不同年齡人羣的不同細胞類型、不同染色體臂的端粒長度數據,或許能以此為基礎,構建出堪比甚至超越甲基化時鐘的端粒時鐘也説不定呢。
不過,目前Telo-seq想要被大規模應用還有一些障礙,比如參考基因組數據的匱乏等。
參考基因組是“實際基因組”的相對概念,代表了某一類物種/組織/細胞系的典型的、健康的基因組結構,參考基因組的構建需要對大量的實際基因組進行彙總、整理、校對才能完成,而有了這樣的基因組作為基準,科學家才好判斷實驗中的實際基因組是什麼樣子。
圖注:參考基因組就像是一個標準化的藍圖,比如成纖維細胞的參考基因組,就為所有實際的成纖維細胞基因組提供了一個共同的參考,如此才能知道實際的成纖維細胞是否發生了某些變異
對於一般的基因而言,參考基因組已經比較完善了,但是針對端粒的參考基因組數據目前還比較有限,想要將Telo-seq用於檢測更多樣化的人類細胞端粒,勢必要豐富相應的參考基因組(該研究使用HG002細胞系也是因為其具備一個公開的高質量參考基因組)。
而在這些“基礎設施”完備之後,衰老檢測體系或將迎來重大更新,在將來,我們或許可以通過這項技術,更直觀地感知到我們身體組織細胞的更新速度、端粒耗損速度等,從而牢牢把握住自己的衰老進程。

端粒縮短與衰老的關係早在上世紀就已經被發現了[7],這種在抗衰界看似“家喻户曉”的概念,我們對其都仍有着如此巨大的認知空白,更別説其他的作為端粒“晚輩”的衰老機制了,只能説我們對衰老的理解,或許仍舊並將長期處於初級階段。
參考文獻
[1] Karimian K, Groot A, Huso V, Kahidi R, Tan K-T, Sholes S, Keener R, McDyer JF, Alder JK, Li H, Rechtsteiner A, Greider CW. Human telomere length is chromosome end-specific and conserved across individuals. Science (New York, NY) 2024; 384:533–539.