AI新模型,助力精準鎖定癌症基因_風聞
中科院之声-中国科学院官方账号-1小时前
癌症的發生與驅動基因突變密切相關,精準識別這些基因是理解癌症機制和開發靶向治療的關鍵。然而,由於癌症成因複雜,現有的基因目錄遠遠不夠完善,現有預測方法在泛化性和可解釋性方面仍存在諸多挑戰。
近日,中國科學院新疆理化技術研究所與合作者,提出了人工智能(AI)可用於癌症驅動基因識別的圖機器學習模型——TREE,通過創新性整合多組學數據,和同質/異質生物網絡拓撲信息,在癌症驅動基因預測的準確性、泛化性和可解釋性方面取得進展。
01
模型突破傳統侷限
識別癌症驅動基因能夠為患者的個性化精準治療提供策略,但至今仍存在大量功能未知、未被標記的癌症基因。
目前,癌症基因預測有兩種主流方法:一是基於多組學的統計方法,二是基於網絡的人工智能方法。雖然兩種方法各有優勢,但也都存在侷限性,比如無法全面捕捉基因間的互動,或者難以處理複雜基因網絡等。
TREE模型的誕生,突破了這些侷限**。**TREE模型是一種基於Transformer的圖表示學習AI模型,能夠處理同質和異質網絡,其中同質網絡僅包含基因,而異質網絡包含轉錄因子(TF)、miRNA和IncRNA等多種節點類型。
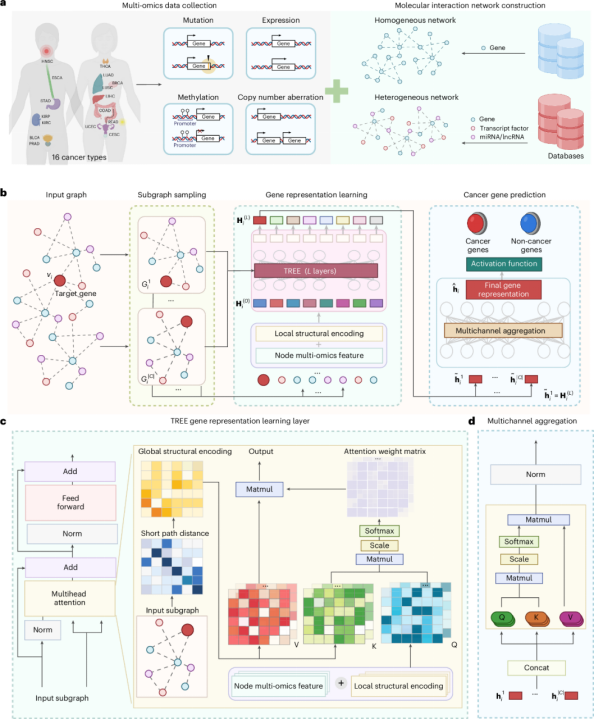
▲(a)多組學數據收集及同質/異質網絡構建;(b)癌症基因預測整體模型流程圖;(c)模型的基因表示學習層;(d)多通道整合模塊。
02
TREE的“超能力”
研究表明,TREE在8個生物泛癌網絡和31個癌症特異性網絡上表現出優越的性能,與5種基於網絡的AI方法相比,TREE的AUC與AUPR指標均表現最佳,平均AUC提升5.91%,AUPR提升9.87%,這都體現出模型的泛化性和魯棒性。
同時,在可解釋性方面,TREE同樣表現出色。突變在癌症基因鑑定中至關重要,而TREE在精確定位罕見突變基因方面具有優勢,異質信息允許TREE通過網絡路徑驗證顯著的癌症基因調控機制。

▲通過所有方法鑑定的癌症候選基因構成的韋恩圖
TREE在對網絡中所有常見的未標記基因進行評分後,推薦了57個潛在的癌症候選基因,認為它們有可能是與癌症相關的候選基因。隨後,科研人員用全部的數據集來測試模型的性能,結果顯示,模型給出的評估結果都很穩定且一致,這表明TREE是識別新的癌症候選基因的可靠工具。
在這57個潛在的癌症候選基因中,有21個也被其他方法鑑定,這些重疊基因獲得了較高的排名。所有這些觀測結果都在相當程度上強調了TREE的可靠性。
03
精準診療“利器”
科研人員選擇了潛在癌症候選基因中的前三名(RYR2,SYNE1和LRP2)進行研究,與相關文獻的結果一致,説明前三名可能參與了癌症的發生和進展。
TREE模型突破了傳統方法在複雜生物網絡解析中的侷限性,為揭示癌症基因的分子調控規律建立了新範式。這種融合多組學分析、圖神經網絡與Transformer架構的跨學科創新,為精準醫療提供了可靠的計算工具。
未來,隨着更多生物數據的積累和算法迭代,TREE有望成為連接基礎研究與臨牀轉化的重要橋樑。其在罕見突變識別、聯合療法靶點發現、癌症早篩等領域的應用潛力,將推動腫瘤診療的發展。
來源**:**中國科學院新疆理化技術研究所