【文獻解讀】CRISPR篩選系統揭示DNA損傷反應中的合成致死關係_風聞
艾迪基因-加速基因编辑进程,造福人类健康1小时前
全面CRISPR篩選揭示DNA修復網絡中的脆弱點
2025年4月,Jacob Corn團隊在《Nature》雜誌發表題為《Comprehensive interrogation of synthetic lethality in the DNA damage response》的研究論文,系統繪製了DNA損傷反應(DNA Damage Response, DDR)核心基因間的遺傳互作圖譜。研究通過構建SPIDR CRISPRi雙向篩選文庫,識別出超過5,000對合成致死基因組合,並深入解析了其中兩對高評分互作的作用機制。這項工作不僅拓展了我們對基因組穩定性維持機制的理解,還為癌症靶向治療提供了新的方向。

01 SPIDR平台:構建覆蓋DNA修復核心基因的系統篩選體系
為全面揭示DDR通路內在的功能重疊與互補機制,研究者設計了一個名為SPIDR(Systematic Profiling of Interactions in DNA Repair)的CRISPRi雙導向文庫,靶向548個核心DDR基因,構建近70萬個sgRNA組合,並在RPE-1細胞中完成篩選。該系統採用CRISPR干擾(CRISPRi)策略避免了Cas9切割引發的DNA損傷,使其適用於研究對細胞生存至關重要的基因組合。
配合GEMINI變分貝葉斯算法對篩選數據進行高通量建模,研究者成功識別出約5,000對合成致死型基因互作(GEMINI score ≤ –1),涵蓋了DNA複製、修復、染色質重塑、轉錄調控等多個生物學過程。SPIDR文庫不僅驗證了多個已知互作(如BRCA2與LIG1),還發現了大量新的功能關聯,為構建細胞穩態下的遺傳互作圖譜提供了堅實工具。
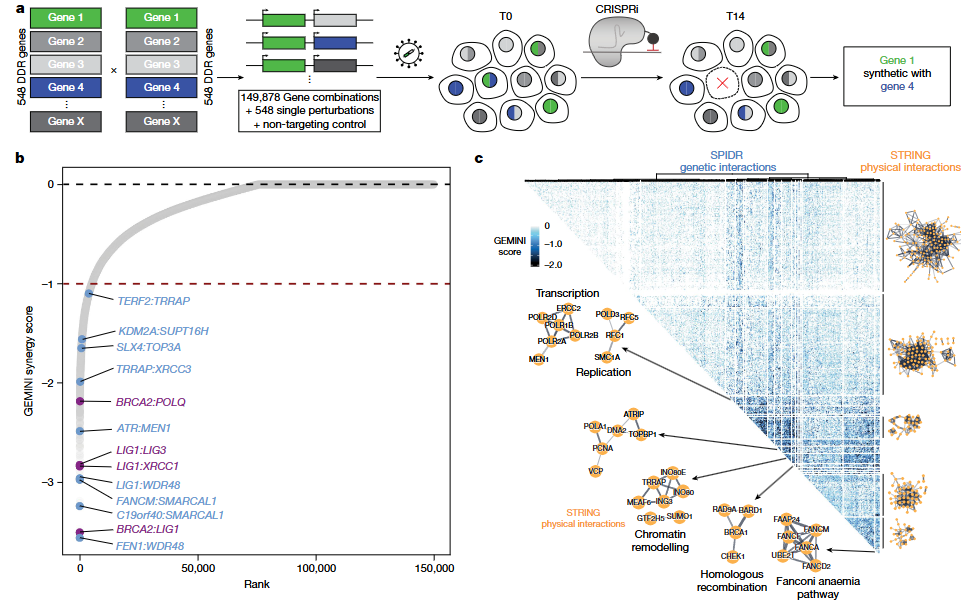
圖1.CRISPR干擾技術對548個核心DNA修復基因進行篩選
02 機制解析:兩對關鍵合成致死組合揭示基因協同維穩機制
在所有發現中,研究團隊聚焦於兩組最具代表性的高評分合成致死互作,解析其在維持基因組完整性中的獨特功能。
其一,FEN1/LIG1 與 WDR48–USP1 的合成致死性源於PCNA泛素化失調。
當FEN1或LIG1缺失時,DNA複製過程中積累的缺口需靠其他機制補救。WDR48–USP1複合物本應去除RAD18介導的PCNA多泛素修飾以維持其穩定性,缺失則導致PCNA降解,引發複製遲滯、染色體斷裂和細胞死亡。研究者還發現,USP1抑制劑對FEN1或LIG1功能低下的細胞具有顯著毒性,提示其潛在的臨牀應用價值。
其二,FANCM 與 SMARCAL1 是兩種DNA轉座酶,主要負責在TA富集區移除潛在形成十字結構(cruciform)的DNA構象。雙缺失會導致這些結構無法及時展開,被ERCC1–ERCC4核酸酶誤切而形成染色體斷裂。該機制不依賴於複製叉逆轉功能,代表了一種新型的結構性基因組損傷源。
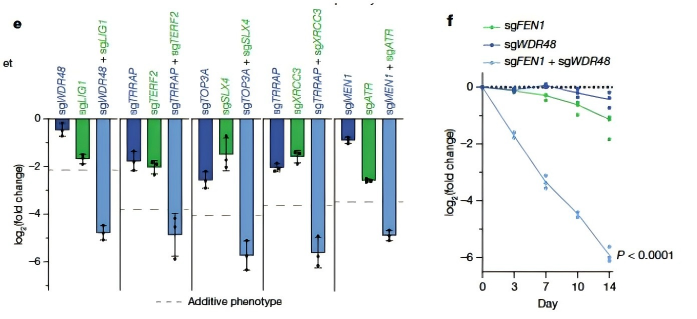
圖2.雙sgRNA競爭生長實驗驗證LIG1:WDR48、FEN1:WDR48的合成致死性
03 從篩選到治療:合成致死網絡驅動精準腫瘤靶點發現
本研究的另一重要意義在於將合成致死互作圖譜與癌症突變數據庫(COSMIC)及藥物靶點數據庫(DGIdb)整合分析,識別出多對理論上可被靶向的小分子-基因組合。例如,攜帶ERCC2突變的膀胱癌細胞可能對DNA-PKcs抑制劑敏感;而FEN1突變型腫瘤可能對USP1抑制劑更為依賴。此外,FANCM在多種癌症中存在突變,提示SMARCAL1為潛在靶向治療因子。
隨着CRISPR技術不斷完善,系統性功能篩選平台如SPIDR將成為發現疾病脆弱性的關鍵手段。尤其在藥物組合設計、耐藥機制分析和個體化治療策略制定中,合成致死圖譜提供了可量化、可驗證的分子基礎。
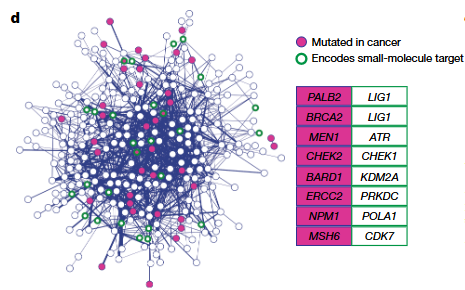
圖3.合成致死網絡圖譜,標註哪些基因對與癌症突變、小分子靶點重疊
總結與展望: CRISPRi篩選開啓功能基因組學精細調控新時代
該研究展示了CRISPRi雙向篩選結合高通量分析平台在解析複雜遺傳網絡中的巨大潛力。通過在無外源壓力條件下揭示合成致死互作,SPIDR平台補足了傳統CRISPR敲除篩選的盲區,並提出多種可轉化的癌症治療策略。未來,類似技術將在腫瘤、神經退行性疾病、免疫系統紊亂等領域中持續拓展其影響力。
艾迪基因專注於CRISPR技術,提供一系列高質量的基因編輯服務和體外診斷產品:CRISPR文庫篩選、細胞基因編輯、單克隆篩選、CRISPR檢測。致力於為CRISPR相關、基因功能研究相關、體外診斷以及治療相關的科學研究提供最高效的技術服務。