衰老“掏空”25%大腦蛋白?大腦衰老真相,或在蛋白質合成的“最後一公里”!_風聞
时光派-时光派官方账号-聚集全球前沿、全面、专业抗衰资讯29分钟前

你是一個在大腦中的平平無奇的核糖體,你的日常就是沿着“信使RNA”的指令帶,把一個個氨基酸串聯成生命所需的蛋白質,一切井然有序。
但隨着衰老,這條路似乎變得異常“粘稠”,常常在特定位置將你卡住,身後不明所以的同伴們繼續撞上來,你們擠作一團,陷入混亂。更詭異的是,大腦似乎對此一無所知,反而下達更多的指令。結果就是,指令越多,合格的產品卻越少,整個大腦處在了無聲的混亂中。
這個現象困擾了生物學界十餘年,根源到底是生產環節出了錯,還是後續的清理環節問題?就在今天,一篇發表於《Science》的重磅研究,最終將矛頭指向了一個具體的分子事件——翻譯過程中的“致命暫停”。


是誰動了我的蛋白質?
所有生命的運作,都遵循着一條神聖的中心法則:DNA先被轉錄為信使RNA(mRNA),mRNA再被核糖體翻譯成蛋白質,去執行細胞的萬千功能。
圖注:分子生物學的中心法則
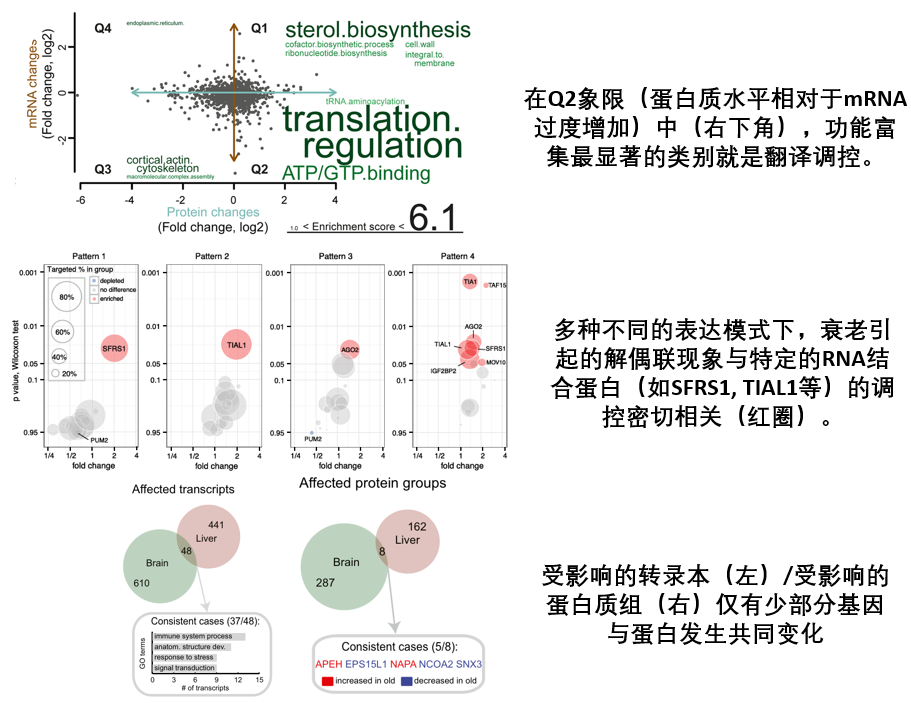
然而,在衰老的大腦中,這條信息鏈似乎出現了“失衡”:指令(mRNA)和最終產品(蛋白質)的數量開始不在同步,二者丰度的變化出現了明顯的不一致,這種“脱鈎”現象,在科學界被稱為“蛋白質—轉錄本解偶聯”。
這個現象並非最近才有的“新瓜”,早在2010年《PLoS Biology》[2]上的一項研究就已經觀察到了類似的情況:衰老的線蟲體內,雖有大量蛋白質發生異常聚集,但它們對應的mRNA指令(總蛋白量)卻並未同步增加,有些甚至還在減少。
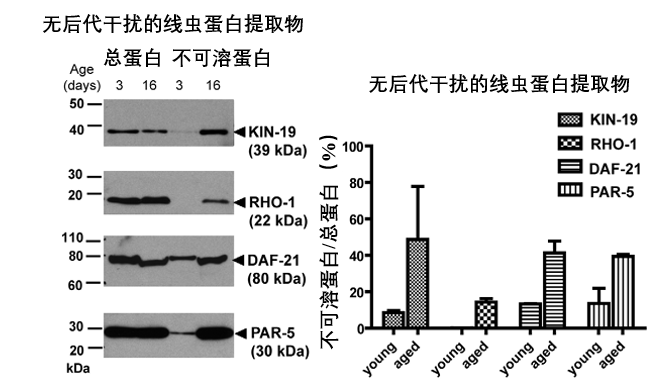
圖注:四種易聚集蛋白的不可溶水平隨衰老而增加,但其總蛋白水平並未增加
隨後,對這一現象的理解在2015年迎來了發展的黃金時期。在此期間,相關的研究成果如雨後春筍般湧現,極大地推動了該領域的發展:從單細胞的酵母[3]、體外培養的人類細胞[4]再到大鼠[5]等多種模型中,科學家們相繼發現了類似的“指令失調”現象。
圖注:酵母衰老的解偶聯特徵(上圖);人類細胞衰老過程中的“解偶聯”現象(中圖);“解偶聯”具有明顯的器官特異性(下圖)
這一系列發現,共同證實“解偶聯”是個貫穿從簡單生物到哺乳動物的、普遍存在的、具有明顯器官特異性的衰老特徵。
現象是有了,但背後的具體機制仍有待研究,目前,整個領域的研究焦點主要集中在兩種可能的主流假説上:一是清理系統(蛋白酶體)隨着衰老效率下降,導致某些蛋白質被異常保留或過度清除[6],或是負責生產蛋白質的核糖體本身出現問題?
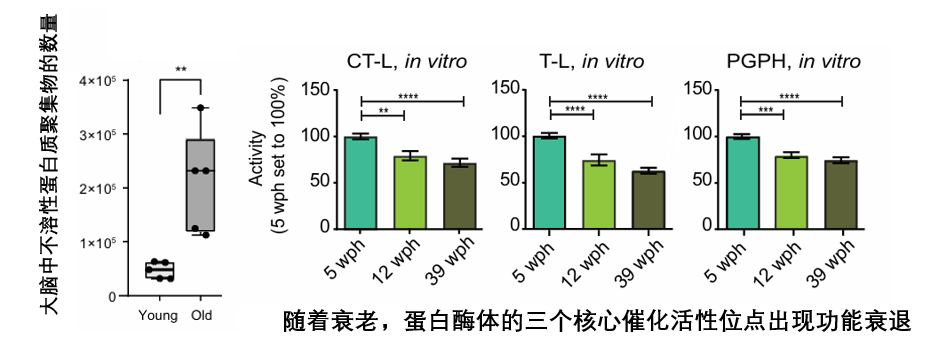
圖注:對於假説一,來自本研究的作者團隊在2020年發現衰老小鼠大腦中蛋白酶體功能下降,是導致部分蛋白質異常的原因之一
二者似乎都有可能發生,那麼,究竟是清理的末端失職,還是生產的源頭就已經出了錯?空談無益,直接用實驗數據來揭曉答案!


致命的暫停
首先是確認並量化“解偶聯”現象。這個簡單,分析了非洲鱂魚從年輕(5周齡)到年老(39周齡)大腦內蛋白質與mRNA的變化後,科學家們發現了“解偶聯”的典型特徵——大量蛋白質的丰度下降,而其對應的mRNA指令卻紋絲不動,甚至有所增加。
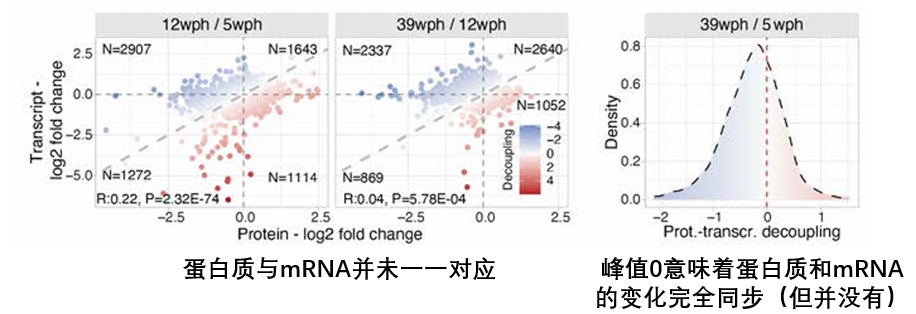
“解偶聯”現象已是板上釘釘,那就可以開始,挨個驗證上面的猜想了!
為了模擬衰老過程中蛋白酶體功能失靈的情況,科學家們特異性地抑制了年輕小魚大腦中的蛋白酶體活性,如果解偶聯的主因真是因為蛋白酶體功能障礙,那麼這種人為模擬所產生的蛋白質變化模式,應該與自然衰老中的模式非常相似(即核糖體蛋白隨着衰老持續減少)。
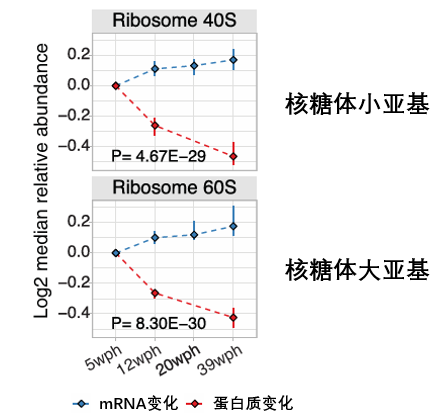
就應該像這樣!
然而,實驗結果給出了一個明確的否定答案:核糖體蛋白的數量不減反增!
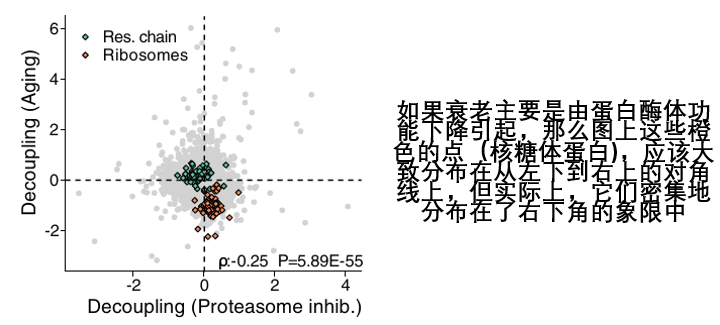
……理論預測與實際結果完全相反,不過這恰恰説明,儘管蛋白酶體功能障礙是衰老過程一部分,但它並不能解釋我們在衰老大腦中看到的核心“解偶聯”現象。
既然清理末端並非主謀,那麼將注意力轉向蛋白質合成本身,便成為了合乎邏輯的下一步,利用核糖體圖譜分析(捕捉細胞內所有正在工作的核糖體,並精確顯示它們在mRNA鏈上的位置),科學家們發現:
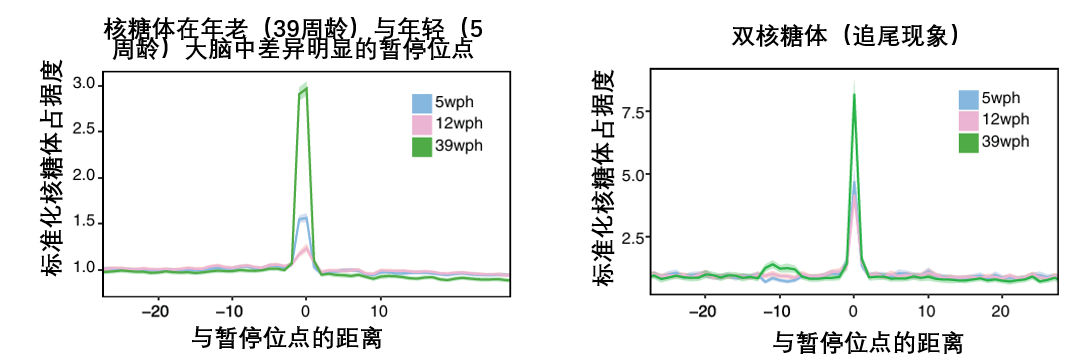
在年老大腦的翻譯過程中,當核糖體在沿着mRNA指令移動時,會在一個個特定的位點上頻繁地出現“卡殼”,發生翻譯延伸暫停現象(下面簡稱翻譯暫停)。
稍微解釋一下,蛋白質的合成包括起始、延伸和終止三個階段,其中,最核心環節是延伸階段,在這個過程中,核糖體會沿着mRNA勻速前進,一個一個地將氨基酸組裝成完整的蛋白質。
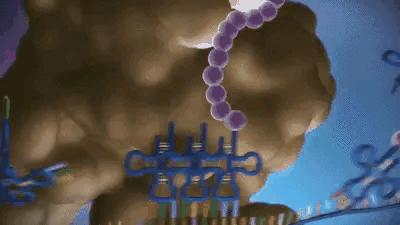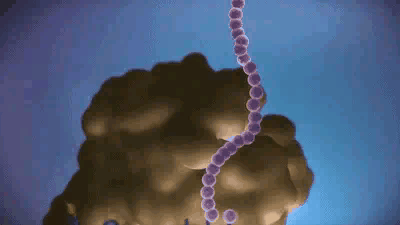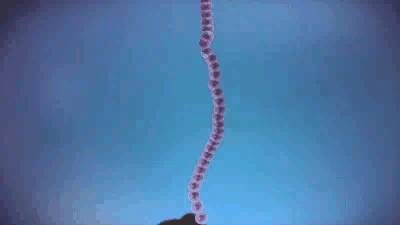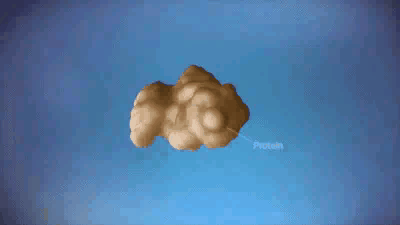
它的速率本應是流暢的(不過並非絕對恆定,適當的減速有時是必要的生理調節)。但在衰老大腦中,這種調節徹底失控,本應流暢移動的核糖體,其前進速率變得極不均勻且明顯變慢,核糖體在特定的位點上發生了長時間的、病理性的停滯。
這種停滯會引發嚴重的連鎖反應,後方正在高速前進的核糖體,會與前方停滯的核糖體發生追尾,導致整條mRNA上的翻譯進程發生擁堵,蛋白質的實際產量遠低於mRNA指令所預示的水平。

細胞似乎也已經感知到了危機,並試圖進行自救——對於那些產量急劇下降的核糖體蛋白,細胞通過延長其mRNA指令的有效期(增加mRNA半衰期)來進行補償,但核糖體已經卡死,再穩定的指令也無濟於事。
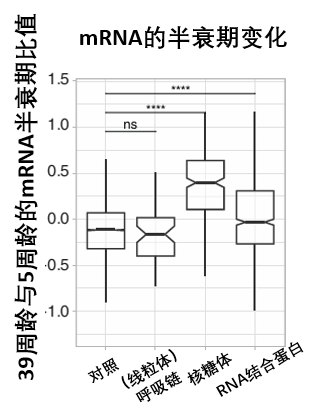
進一步的分析表明,這種暫停並非“隨地大小停”,而是高發於編碼兩種特定氨基酸——賴氨酸(Lysine,K)和精氨酸(Arginine,R)的密碼子序列上,這兩種氨基酸都有個共同點:帶有正電荷(下圖藍色部分)。

但為什麼偏偏是它們?這是因為蛋白質是在一條被稱為“核糖體出口通道”的狹長隧道中被合成並擠出來的。這條隧道的內壁,本身是帶有負電荷的,所以帶正電這倆氨基酸經過時,就會產生阻力,減慢翻譯延伸的速率[7]。
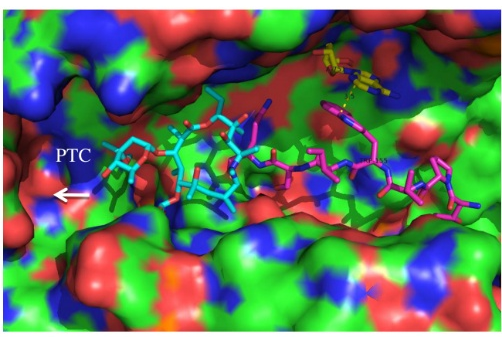
圖注:核糖體出口通道的內部視圖(箭頭所指為出口方向)
而問題的嚴重性在於,細胞內的核心蛋白質,恰恰就是這些正電氨基酸的“富集區”。 無論是負責維持基因組穩定和執行基因指令的RNA結合蛋白,還是關鍵的DNA修復蛋白,它們的結構和功能實現……都高度依賴於序列中包含大量的賴氨酸和精氨酸。
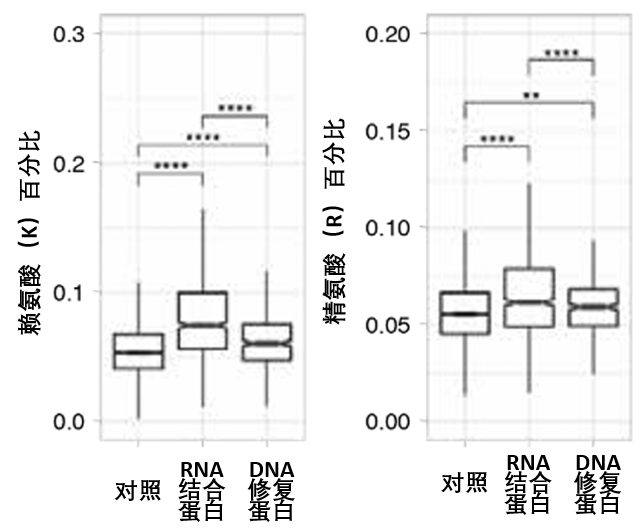
因此,當衰老引發的翻譯暫停,精準地發生在這些富含鹼性氨基酸的序列上時,其後果顯然是極其災難性的。如果某一特定類型的神經元,其功能高度依賴於這些富含賴氨酸和精氨酸的關鍵蛋白質,那麼它們在衰老過程中,自然會成為最先崩潰的“薄弱環節”。
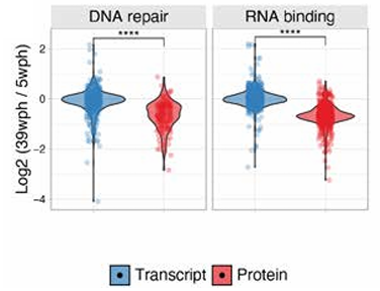
圖注:隨着衰老,負責RNA結合與DNA修復的核心蛋白質,其數量都出現了下降(紅),儘管它們的mRNA並未減少(藍)

未解的根源
上面已經明確,衰老大腦中的核糖體會在特定位點發生翻譯暫停,但還有個問題亟待解決:是什麼原因,導致了這場現象的發生?
順着這條線索往下查,研究團隊很快將目光投向了蛋白質合成環節的搬運工之一:轉運RNA(tRNA)。
在分析了它攜帶氨基酸時的活性狀態與自身丰度後,結果表明,雖然衰老大腦中tRNA攜帶氨基酸的整體效率確實有所下降,而且這種下降的模式,與翻譯暫停發生的具體位點特異性……貌似並不能完全對應。
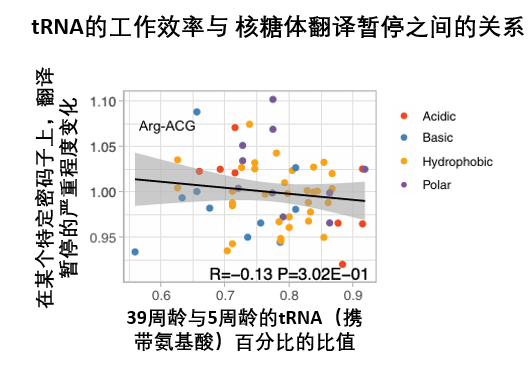
圖注:tRNA供應的改變很可能只是導致翻譯暫停的因素之一,並不是根本原因或唯一原因。
所以,根本原因是否在於核糖體本身在衰老過程中發生了不可逆的化學損傷?或是mRNA在衰老過程中出現了某些化學修飾,從而影響其被翻譯的流暢度?細胞能量狀態的改變,同樣可能在其中扮演了關鍵角色,這都有可能,目前還未可知……

時光派點評
儘管目前仍有許多未解之謎,但這項研究,為我們理解神經退行性疾病以及詮釋“衰老標識”的內涵,提供了全新的解釋角度。
神經退行性疾病的新角度
長期以來,神經退行性疾病的一個核心研究焦點在於其病理結果——特定蛋白質的錯誤摺疊與聚集。這也是許多主流藥物研發的研發思路,即如何有效清除這些這些蛋白質垃圾?
讀完本文,你或許就能意識到,這些疾病的根源,或許不在於“清理不利”,而在於“生產出錯”:衰老引發的翻譯暫停和碰撞,本身就會源源不斷地製造出大量不完整、結構異常的蛋白質半成品,這或許才是催生出那些“老年斑”或“神經纖維纏結”的真正源頭。
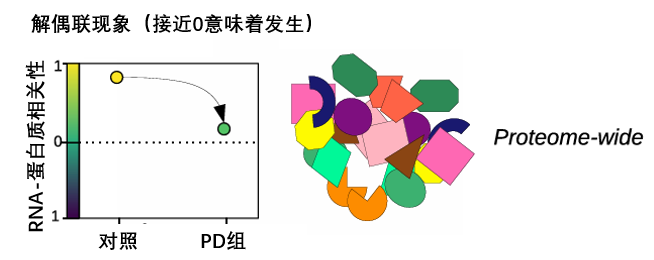
圖注:在帕金森病患者的大腦中,也已經觀察到了顯著的“解偶聯”現象[8]
衰老標識的上游?
過去,衰老通常被看作是多個“衰老標誌”的集合,這些標誌間影響錯綜複雜。這篇研究揭示:多個關鍵標識(如基因組不穩定性和蛋白質穩態喪失等),可能受到同一個上游的事件驅動——那些富含鹼性氨基酸(賴氨酸、精氨酸)的核心蛋白質的合成受阻。
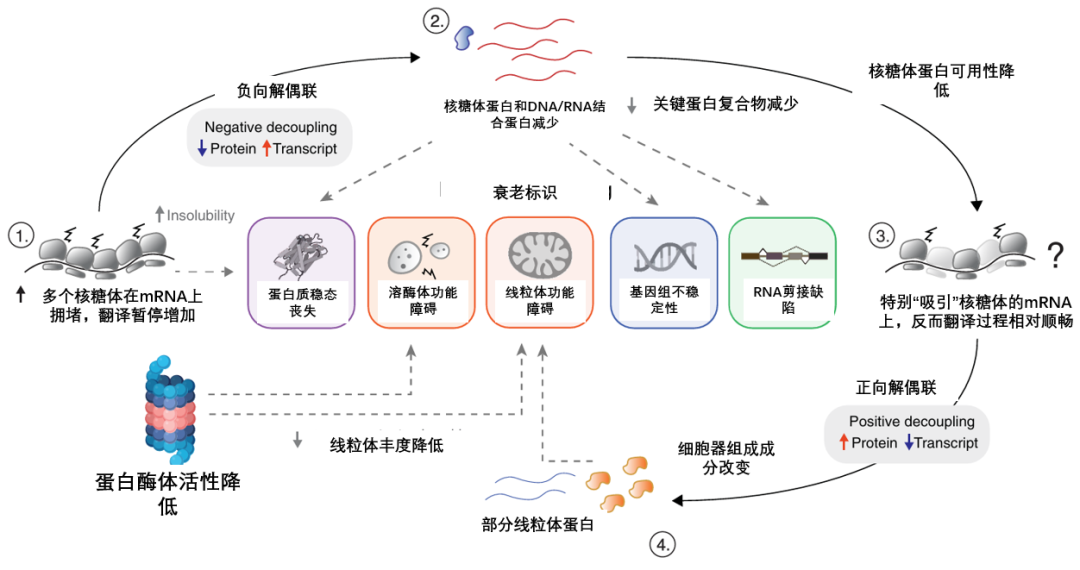
圖注:還有正向解偶聯(右下角):由於核糖體總數下降,對於少數能高效啓動翻譯的mRNA(如部分線粒體蛋白的轉錄本),反而因為擁堵緩解,其蛋白質產量不降反升,但它會破壞線粒體的內部平衡,加劇“線粒體功能障礙”
這意味着,每當出現翻譯暫停時,它最先影響的,正是那些對細胞來説最關鍵的核心蛋白!就比如DNA修復蛋白的減少,引發了“基因組不穩定”;而核糖體蛋白自身的減少,則動搖了整個大分子生物合成的根基。
或許,維持蛋白質合成路線的暢通,才是從根本上延緩衰老的關鍵所在。
聲明 - 本文內容僅用於科普知識分享與抗衰資訊傳遞,不構成對任何產品、技術或觀點的推薦、背書或功效證明。文內提及效果僅指成分特性,非疾病治療功能。涉及健康、醫療、科技應用等相關內容僅供參考,醫療相關請尋求專業醫療機構並遵醫囑,本文不做任何醫療建議。如欲轉載本文,請與本公眾號聯繫授權與轉載規範。
參考文獻
[1] https://doi.org/10.1126/science.adk3079
[2] David DC, Ollikainen N, Trinidad JC, Cary MP, Burlingame AL, Kenyon C. Widespread protein aggregation as an inherent part of aging in C. elegans. PLoS Biol. 2010 Aug 10;8(8):e1000450. doi: 10.1371/journal.pbio.1000450. PMID: 20711477; PMCID: PMC2919420.
[3] Janssens GE, Meinema AC, González J, Wolters JC, Schmidt A, Guryev V, Bischoff R, Wit EC, Veenhoff LM, Heinemann M. Protein biogenesis machinery is a driver of replicative aging in yeast. Elife. 2015 Dec 1;4:e08527. doi: 10.7554/eLife.08527. PMID: 26422514; PMCID: PMC4718733.
[4] Wei YN, Hu HY, Xie GC, Fu N, Ning ZB, Zeng R, Khaitovich P. Transcript and protein expression decoupling reveals RNA binding proteins and miRNAs as potential modulators of human aging. Genome Biol. 2015 Feb 22;16(1):41. doi: 10.1186/s13059-015-0608-2. PMID: 25853883; PMCID: PMC4375924.
[5] Ori A, Toyama BH, Harris MS, Bock T, Iskar M, Bork P, Ingolia NT, Hetzer MW, Beck M. Integrated Transcriptome and Proteome Analyses Reveal Organ-Specific Proteome Deterioration in Old Rats. Cell Syst. 2015 Sep 23;1(3):224-37. doi: 10.1016/j.cels.2015.08.012. Epub 2015 Sep 17. PMID: 27135913; PMCID: PMC4802414.
[6] Kelmer Sacramento E, Kirkpatrick JM, Mazzetto M, Baumgart M, Bartolome A, Di Sanzo S, Caterino C, Sanguanini M, Papaevgeniou N, Lefaki M, Childs D, Bagnoli S, Terzibasi Tozzini E, Di Fraia D, Romanov N, Sudmant PH, Huber W, Chondrogianni N, Vendruscolo M, Cellerino A, Ori A. Reduced proteasome activity in the aging brain results in ribosome stoichiometry loss and aggregation. Mol Syst Biol. 2020 Jun;16(6):e9596. doi: 10.15252/msb.20209596. PMID: 32558274; PMCID: PMC7301280.
[7] Llewellyn J, Hubbard SJ, Swift J. Translation is an emerging constraint on protein homeostasis in ageing. Trends Cell Biol. 2024 Aug;34(8):646-656. doi: 10.1016/j.tcb.2024.02.001. Epub 2024 Feb 28. PMID: 38423854.
[8] Dick F, Tysnes OB, Alves GW, Nido GS, Tzoulis C. Altered transcriptome-proteome coupling indicates aberrant proteostasis in Parkinson’s disease. iScience. 2023 Jan 4;26(2):105925. doi: 10.1016/j.isci.2023.105925. PMID: 36711240; PMCID: PMC9874017.