阿里雲生物基礎大模型登上Nature子刊,可挖掘核酸、蛋白質之間的內在聯繫
【環球網科技綜合報道】6月19日消息,國際頂級期刊《Nature Machine Intelligence》發表了阿里雲AI for Science的研究成果LucaOne。這是業界首個聯合DNA、RNA、蛋白質的生物大模型。該大模型學習了超16萬個物種的12億條核酸序列及6億條蛋白序列數據,不僅可以挖掘核酸、蛋白質的內部特徵,還可識別核酸與蛋白質之間的聯繫,幫助研究人員探索更多生物系統的內在邏輯與規則。
Nature Machine Intelligence(以下簡稱NMI)是Nature於2019年創立的期刊,收錄方向覆蓋計算生物學、模式識別和計算機視覺等多個領域,期刊影響因子一直位居行業前列,是業界公認的人工智能和機器學習領域的國際頂級期刊。
據介紹,阿里雲研究團隊首次基於DNA、RNA和蛋白質等生命科學領域最主要的數據進行混合訓練,涵蓋12億條核酸序列和6億條蛋白序列,同時引入生物領域內基礎的標籤信息,讓模型學習到豐富的生物信息。在模型結構上,研究團隊採用Transformer-Encoder架構,在自監督學習的基礎上,設計了8個不同級別的半監督學習任務,有效增強模型的學習能力。
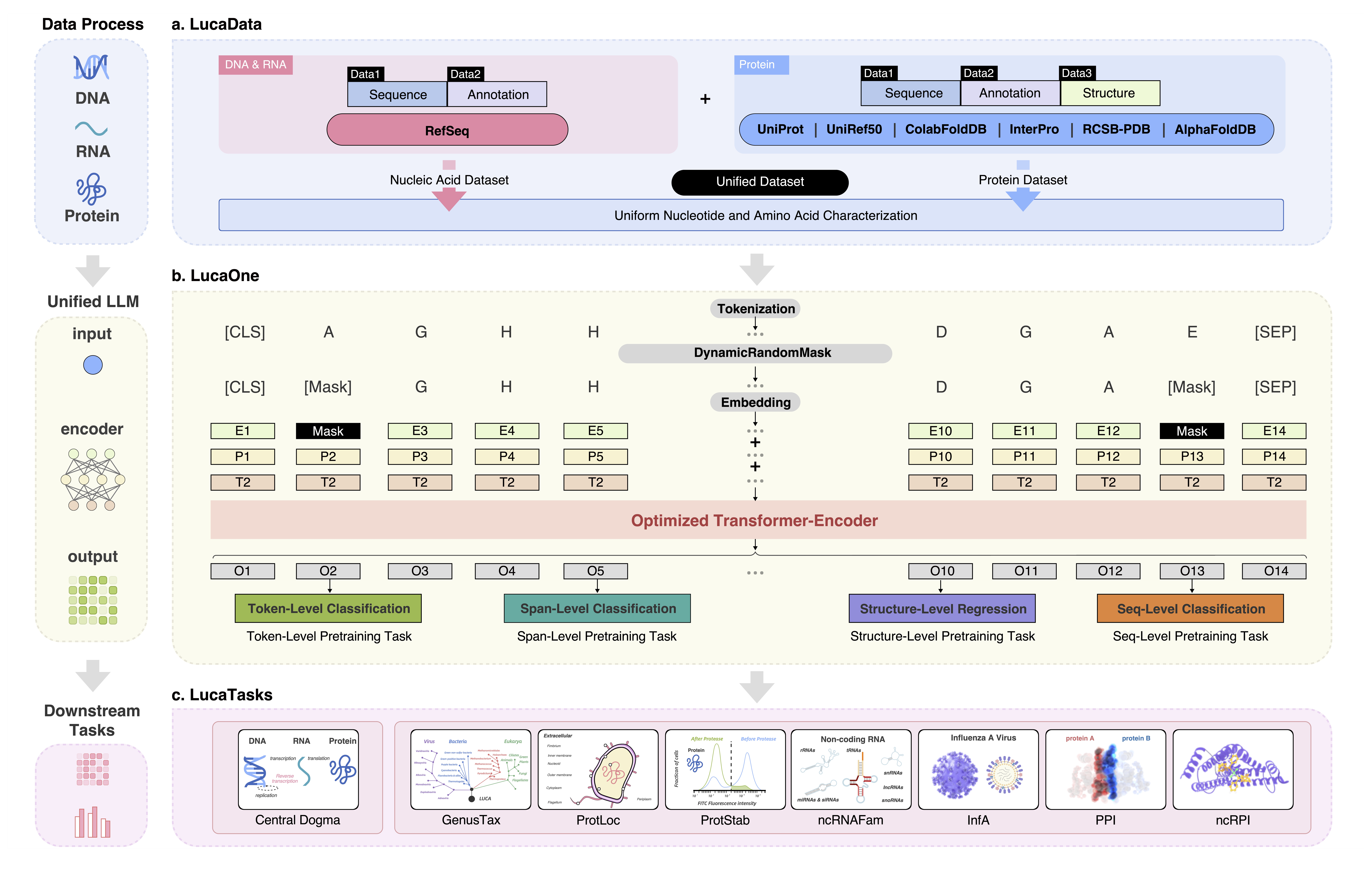
實驗結果顯示,在中心法則驗證(CentralDogma)、物種Genus分類(GenusTax)、蛋白質位置(ProtLoc)、蛋白質穩定性(ProtStab)、非編碼RNA的類型(ncRNAFam)、流感病毒預測(InfA)等8個下游任務驗證中,LucaOne的表現均領先於現有的生物大模型。
NMI表示:該研究展示了對分子生物學中心法則的全新理解,極大地增強了研究人員對生物信息學分析的能力,可以幫助人類探索分子生物學的未知領域。
據介紹,LucaOne的模型代碼、訓練代碼、推理代碼等已全面開源,科研人員可基於該模型進行二次訓練、垂直領域生物模型構建、Embedding推理、Embedding-based分析、Embedding-based下游模型構建等。目前全球已有10多家公司和團隊使用該模型。
過去幾年,阿里雲積極與國內高校和研究機構展開合作,在生命科學領域已發表核酸和蛋白質統一基礎模型-LucaOne(NMI 2025)、RNA病毒發現-LucaProt(Cell 2024)、磷循環蛋白家族識別-LucaPCycle(NC 2025)等研究成果。(勃潺)